8 Ingeniería De Características Con recipes
La ingeniería de características implica reformatear los valores de los predictores para que sea más fácil de usar para un modelo de manera efectiva. Esto incluye transformaciones y codificaciones de los datos para representar mejor sus características importantes. Imagine que tiene dos predictores en un conjunto de datos que pueden representarse más eficazmente en su modelo como una proporción; crear un nuevo predictor a partir de la proporción de los dos originales es un ejemplo simple de ingeniería de características.
Tomemos como ejemplo la ubicación de una casa en Ames. Hay diversas formas en que esta información espacial puede exponerse a un modelo, incluida la vecindad (una medida cualitativa), la longitud/latitud, la distancia a la escuela más cercana o a la Universidad Estatal de Iowa, etc. Al elegir cómo codificar estos datos en el modelado, podemos elegir una opción que creamos que esté más asociada con el resultado. El formato original de los datos, por ejemplo numérico (por ejemplo, distancia) versus categórico (por ejemplo, vecindad), también es un factor determinante en las elecciones de ingeniería de características.
Otros ejemplos de preprocesamiento para crear mejores funciones para el modelado incluyen:
La correlación entre predictores se puede reducir mediante la extracción de características o la eliminación de algunos predictores.
Cuando a algunos predictores les faltan valores, se pueden imputar mediante un submodelo.
Los modelos que utilizan medidas de tipo varianza pueden beneficiarse al forzar la distribución de algunos predictores sesgados para que sean simétricas mediante la estimación de una transformación.
La ingeniería de funciones y el preprocesamiento de datos también pueden implicar el reformateo que puede requerir el modelo. Algunos modelos utilizan métricas de distancia geométricas y, en consecuencia, los predictores numéricos deben centrarse y escalarse para que estén todos en las mismas unidades. De lo contrario, los valores de distancia estarían sesgados por la escala de cada columna.
Los diferentes modelos tienen diferentes requisitos de preprocesamiento y algunos, como los modelos basados en árboles, requieren muy poco preprocesamiento. El Apéndice A contiene una pequeña tabla de técnicas de preprocesamiento recomendadas para diferentes modelos.
En este capítulo, presentamos el paquete recipes que puede usar para combinar diferentes tareas de ingeniería de características y preprocesamiento en un solo objeto y luego aplicar estas transformaciones. a diferentes conjuntos de datos. El paquete recipes es, como parsnip para los modelos, uno de los paquetes principales de tidymodels.
Este capítulo utiliza los datos de alojamiento de Ames y los objetos R creados en el libro hasta ahora, como se resume en Sección 7.7.
8.1 Una recipe() Simple Para Los Datos De Vivienda De Ames
En esta sección, nos centraremos en un pequeño subconjunto de los predictores disponibles en los datos de vivienda de Ames:
El vecindario (cualitativo, con vecindarios 29 en el conjunto de entrenamiento)
La superficie habitable bruta sobre el nivel del suelo (continua, denominada
Gr_Liv_Area)El año de construcción (
Year_Built)El tipo de edificio (
Bldg_Typecon valoresOneFam(\(n = 1,936\)),TwoFmCon(\(n = 50\)),Duplex(\(n = 88\)),Twnhs(\(n = 77\)), andTwnhsE(\(n = 191\)))
Supongamos que se ajustara un modelo de regresión lineal ordinaria inicial a estos datos. Recordando que, en el Capítulo 4, los precios de venta estaban registrados previamente, una llamada estándar a lm() podría verse así:
Cuando se ejecuta esta función, los datos se convierten de un marco de datos a una matriz de diseño numérica (también llamada matriz de modelo) y luego se utiliza el método de mínimos cuadrados para estimar los parámetros. En Sección 3.2 enumeramos los múltiples propósitos de la fórmula del modelo R; Centrémonos sólo en los aspectos de manipulación de datos por ahora. Lo que hace esta fórmula se puede descomponer en una serie de pasos:
El precio de venta se define como el resultado, mientras que las variables de vecindario, superficie habitable bruta, año de construcción y tipo de edificio se definen como predictores.
Se aplica una transformación logarítmica al predictor de superficie habitable bruta.
Las columnas de vecindario y tipo de edificio se convierten de un formato no numérico a un formato numérico (ya que los mínimos cuadrados requieren predictores numéricos).
As mentioned in Chapter 3, the formula method will apply these data manipulations to any data, including new data, that are passed to the predict() function.
Una receta también es un objeto que define una serie de pasos para el procesamiento de datos. A diferencia del método de fórmula dentro de una función de modelado, la receta define los pasos mediante funciones step_*() sin ejecutarlas inmediatamente; es sólo una especificación de lo que se debe hacer. Aquí hay una receta equivalente a la fórmula anterior que se basa en el resumen del código en Sección 5.5:
library(tidymodels) # Incluye el paquete de recetas.
tidymodels_prefer()
simple_ames <-
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type,
data = ames_train) %>%
step_log(Gr_Liv_Area, base = 10) %>%
step_dummy(all_nominal_predictors())
simple_ames
##
## ── Recipe ───────────────────────────────────────────────────────────────────────────
##
## ── Inputs
## Number of variables by role
## outcome: 1
## predictor: 4
##
## ── Operations
## • Log transformation on: Gr_Liv_Area
## • Dummy variables from: all_nominal_predictors()Analicemos esto:
La llamada a
recipe()(recipe es receta en español) con una fórmula le dice a la receta las funciones de los “ingredientes” o variables (por ejemplo, predictor, resultado). Solo utiliza los datosames_trainpara determinar los tipos de datos de las columnas.step_log()declara queGr_Liv_Areadebe transformarse en registros.step_dummy()especifica qué variables deben convertirse de un formato cualitativo a un formato cuantitativo, en este caso, utilizando variables ficticias o indicadoras. Un indicador o variable ficticia es una variable numérica binaria (una columna de unos y ceros) que codifica información cualitativa; Profundizaremos en este tipo de variables en Sección 8.4.1.
La función all_nominal_predictors() captura los nombres de cualquier columna predictora que actualmente sea de naturaleza factor o carácter (es decir, nominal). Esta es una función selectora similar a dplyr similar a starts_with() o matches() pero que solo se puede usar dentro de una receta.
Otros selectores específicos del paquete recipes son: all_numeric_predictors(), all_numeric(), all_predictors() y all_outcomes(). Al igual que con dplyr, se pueden usar una o más expresiones sin comillas, separadas por comas, para seleccionar qué columnas se ven afectadas por cada paso.
¿Cuál es la ventaja de utilizar una receta sobre una fórmula o predictores sin procesar? Hay algunos, que incluyen:
Estos cálculos se pueden reciclar entre modelos, ya que no están estrechamente vinculados a la función de modelado.
Una receta permite un conjunto más amplio de opciones de procesamiento de datos que las que pueden ofrecer las fórmulas.
La sintaxis puede ser muy compacta. Por ejemplo,
all_nominal_predictors()se puede utilizar para capturar muchas variables para tipos específicos de procesamiento, mientras que una fórmula requeriría que cada una de ellas se enumere explícitamente.Todo el procesamiento de datos se puede capturar en un único objeto R en lugar de en scripts que se repiten o incluso se distribuyen en diferentes archivos.
8.2 Usando Recetas
Como comentamos en el Capítulo 7, las opciones de preprocesamiento y la ingeniería de características normalmente deben considerarse parte de un flujo de trabajo de modelado, no una tarea separada. El paquete workflows contiene funciones de alto nivel para manejar diferentes tipos de preprocesadores. Nuestro flujo de trabajo anterior (lm_wflow) usaba un conjunto simple de selectores dplyr. Para mejorar ese enfoque con ingeniería de características más compleja, usemos la receta simple_ames para preprocesar datos para modelar.
Este objeto se puede adjuntar al flujo de trabajo:
lm_wflow %>%
add_recipe(simple_ames)
## Error in `add_recipe()`:
## ! A recipe cannot be added when variables already exist.¡Eso no funcionó! Solo podemos tener un método de preprocesamiento a la vez, por lo que debemos eliminar el preprocesador existente antes de agregar la receta.
lm_wflow <-
lm_wflow %>%
remove_variables() %>%
add_recipe(simple_ames)
lm_wflow
## ══ Workflow ═════════════════════════════════════════════════════════════════════════
## Preprocessor: Recipe
## Model: linear_reg()
##
## ── Preprocessor ─────────────────────────────────────────────────────────────────────
## 2 Recipe Steps
##
## • step_log()
## • step_dummy()
##
## ── Model ────────────────────────────────────────────────────────────────────────────
## Linear Regression Model Specification (regression)
##
## Computational engine: lmEstimemos tanto la receta como el modelo usando una simple llamada a fit():
lm_fit <- fit(lm_wflow, ames_train)El método predict() aplica el mismo preprocesamiento que se usó en el conjunto de entrenamiento a los nuevos datos antes de pasarlos al método predict() del modelo:
predict(lm_fit, ames_test %>% slice(1:3))
## # A tibble: 3 × 1
## .pred
## <dbl>
## 1 5.08
## 2 5.32
## 3 5.28Si necesitamos el objeto del modelo básico o la receta, existen funciones extract_* que pueden recuperarlos:
# Obtén la receta una vez estimada:
lm_fit %>%
extract_recipe(estimated = TRUE)
##
## ── Recipe ───────────────────────────────────────────────────────────────────────────
##
## ── Inputs
## Number of variables by role
## outcome: 1
## predictor: 4
##
## ── Training information
## Training data contained 2342 data points and no incomplete rows.
##
## ── Operations
## • Log transformation on: Gr_Liv_Area | Trained
## • Dummy variables from: Neighborhood and Bldg_Type | Trained
# Para ordenar el ajuste del modelo:
lm_fit %>%
# Esto devuelve el objeto parsnip:
extract_fit_parsnip() %>%
# Ahora ordena el objeto del modelo lineal:
tidy() %>%
slice(1:5)
## # A tibble: 5 × 5
## term estimate std.error statistic p.value
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 (Intercept) -0.669 0.231 -2.90 3.80e- 3
## 2 Gr_Liv_Area 0.620 0.0143 43.2 2.63e-299
## 3 Year_Built 0.00200 0.000117 17.1 6.16e- 62
## 4 Neighborhood_College_Creek 0.0178 0.00819 2.17 3.02e- 2
## 5 Neighborhood_Old_Town -0.0330 0.00838 -3.93 8.66e- 5Las herramientas para usar (y depurar) recetas fuera de los objetos del flujo de trabajo se describen en Sección 16.4.
8.3 Cómo Se Utilizan Los Datos En recipe()
Los datos se pasan a recetas en diferentes etapas.
Primero, al llamar a recipe(..., data), el conjunto de datos se usa para determinar los tipos de datos de cada columna, de modo que se puedan usar selectores como all_numeric() o all_numeric_predictors().
En segundo lugar, cuando se preparan los datos usando “fit(workflow, data)”, los datos de entrenamiento se utilizan para todas las operaciones de estimación, incluida una receta que puede ser parte del “workflow”, desde determinar los niveles de los factores hasta calcular los componentes de PCA y todo lo demás.
Todos los pasos de preprocesamiento e ingeniería de funciones utilizan solo los datos de entrenamiento. De lo contrario, la fuga de información puede afectar negativamente el rendimiento del modelo cuando se utiliza con datos nuevos.
Finalmente, cuando se usa predict(workflow, new_data), ningún modelo o parámetro de preprocesador como los de las recetas se reestima usando los valores en new_data. Tome el centrado y el escalado usando step_normalize() como ejemplo. Con este paso, las medias y las desviaciones estándar de las columnas apropiadas se determinan a partir del conjunto de entrenamiento; Las nuevas muestras en el momento de la predicción se estandarizan utilizando estos valores del entrenamiento cuando se invoca predict().
8.4 Ejemplos De Pasos De Recetas
Antes de continuar, hagamos un recorrido extenso por las capacidades de recipes y exploremos algunas de las funciones más importantes de step_*(). Cada una de estas funciones de pasos de recetas especifica un posible paso específico en un proceso de ingeniería de características, y diferentes pasos de recetas pueden tener diferentes efectos en las columnas de datos.
8.4.1 Codificación de datos cualitativos en formato numérico
Una de las tareas de ingeniería de características más comunes es transformar datos nominales o cualitativos (factores o caracteres) para que puedan codificarse o representarse numéricamente. A veces podemos alterar los niveles de factores de una columna cualitativa de manera útil antes de dicha transformación. Por ejemplo, step_unknown() se puede utilizar para cambiar los valores faltantes a un nivel de factor dedicado. De manera similar, si anticipamos que se puede encontrar un nuevo nivel de factor en datos futuros, step_novel() puede asignar un nuevo nivel para este propósito.
Además, step_other() se puede utilizar para analizar las frecuencias de los niveles de factores en el conjunto de entrenamiento y convertir valores que ocurren con poca frecuencia a un nivel general de “otro”, con un umbral que se puede especificar. Un buen ejemplo es el predictor Neighborhood de nuestros datos, que se muestra en Figura 8.1.
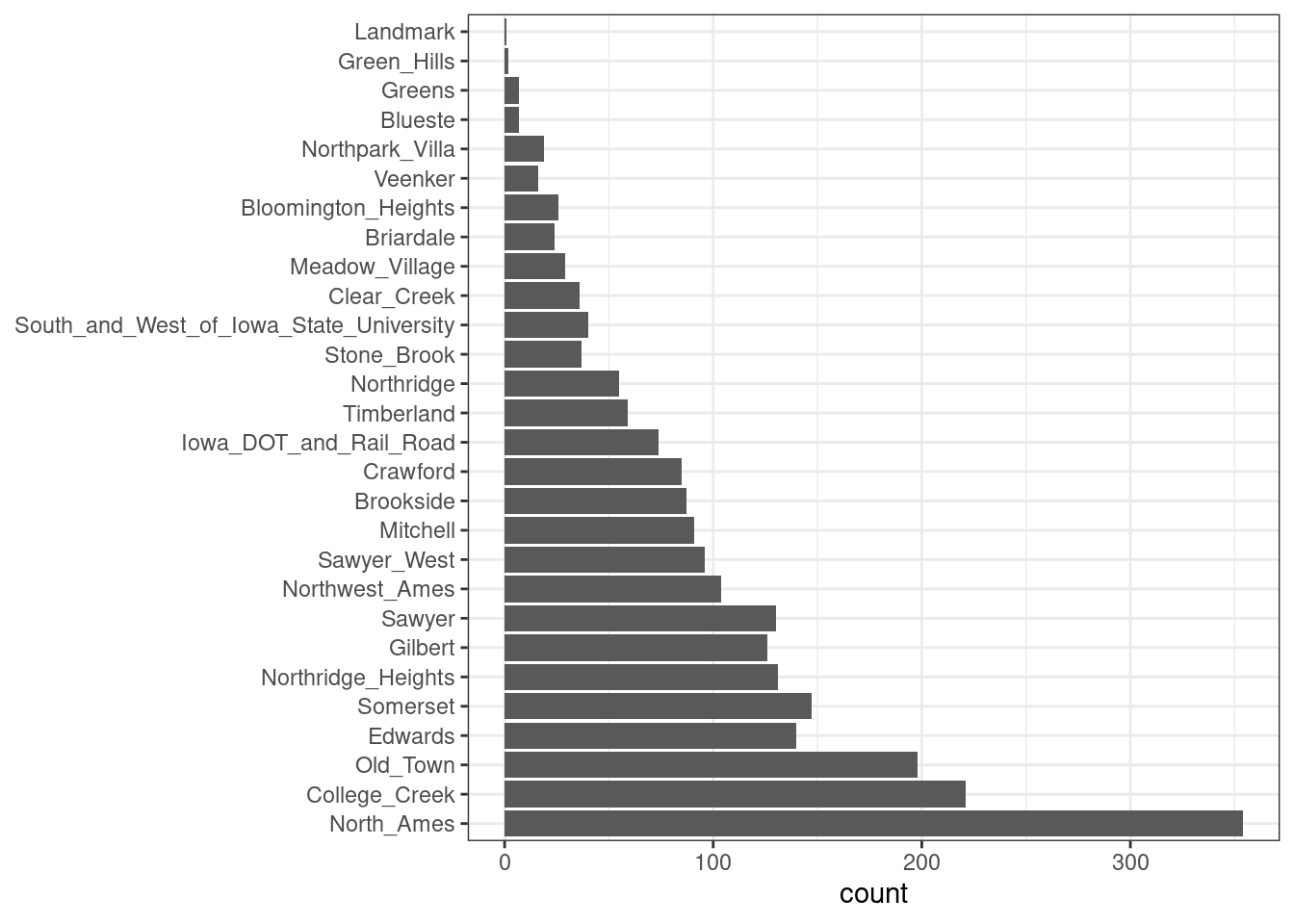
Aquí vemos que dos vecindarios tienen menos de cinco propiedades en los datos de entrenamiento (Landmark y Green Hills); en este caso, no se incluyó ninguna casa en el vecindario Landmark en el conjunto de pruebas. Para algunos modelos, puede resultar problemático tener variables ficticias con una única entrada distinta de cero en la columna. Como mínimo, es muy improbable que estas características sean importantes para un modelo. Si agregamos step_other(Neighborhood, umbral = 0.01) a nuestra receta, el 1% inferior de los vecindarios se agrupará en un nuevo nivel llamado “otros”. En este conjunto de entrenamiento, esto capturará los vecindarios seven.
Para los datos de Ames, podemos modificar la receta para usar:
simple_ames <-
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type,
data = ames_train) %>%
step_log(Gr_Liv_Area, base = 10) %>%
step_other(Neighborhood, threshold = 0.01) %>%
step_dummy(all_nominal_predictors())Muchos, pero no todos, los cálculos del modelo subyacente requieren que los valores predictores se codifiquen como números. Las excepciones notables incluyen modelos basados en árboles, modelos basados en reglas y modelos ingenuos de Bayes.
El método más común para convertir un factor predictivo a un formato numérico es crear variables ficticias o indicadoras. Tomemos el predictor en los datos de Ames para el tipo de edificio, que es una variable factorial con cinco niveles (ver Tabla 8.1). Para variables ficticias, la única columna Bldg_Type se reemplazaría con cuatro columnas numéricas cuyos valores son cero o uno. Estas variables binarias representan valores de nivel de factor específicos. En R, la convención es excluir una columna para el primer nivel de factor (OneFam, en este caso). La columna Bldg_Type se reemplazaría con una columna llamada TwoFmCon que es uno cuando la fila tiene ese valor y cero en caso contrario. Se crean otras tres columnas de manera similar:
| Raw Data | TwoFmCon | Duplex | Twnhs | TwnhsE |
|---|---|---|---|---|
| OneFam | 0 | 0 | 0 | 0 |
| TwoFmCon | 1 | 0 | 0 | 0 |
| Duplex | 0 | 1 | 0 | 0 |
| Twnhs | 0 | 0 | 1 | 0 |
| TwnhsE | 0 | 0 | 0 | 1 |
¿Por qué no los cinco? La razón más básica es la simplicidad; Si conoce el valor de estas cuatro columnas, puede determinar el último valor porque son categorías mutuamente excluyentes. Más técnicamente, la justificación clásica es que varios modelos, incluida la regresión lineal ordinaria, tienen problemas numéricos cuando existen dependencias lineales entre columnas. Si se incluyen las cinco columnas de indicadores de tipo de edificio, se sumarían a la columna de intersección (si hay una). Esto causaría un problema, o quizás un error total, en el álgebra matricial subyacente.
El conjunto completo de codificaciones se puede utilizar para algunos modelos. Esto se denomina tradicionalmente codificación one-hot y se puede lograr utilizando el argumento one_hot de step_dummy().
Una característica útil de step_dummy() es que hay más control sobre cómo se nombran las variables ficticias resultantes. En base R, los nombres de variables ficticias combinan el nombre de la variable con el nivel, lo que da como resultado nombres como NeighborhoodVeenker. Las recetas, de forma predeterminada, usan un guión bajo como separador entre el nombre y el nivel (por ejemplo, Neighborhood_Veenker) y existe una opción para usar formato personalizado para los nombres. La convención de nomenclatura predeterminada en recipes hace que sea más fácil capturar esas nuevas columnas en pasos futuros usando un selector, como starts_with("Neighborhood_").
Las variables ficticias tradicionales requieren que se conozcan todas las categorías posibles para crear un conjunto completo de características numéricas. Existen otros métodos para realizar esta transformación a un formato numérico. Los métodos de hashing de características solo consideran el valor de la categoría para asignarlo a un grupo predefinido de variables ficticias. Las codificaciones de efecto o probabilidad reemplazan los datos originales con una única columna numérica que mide el efecto de esos datos. Tanto el hash de características como la codificación de efectos pueden manejar sin problemas situaciones en las que se encuentra un nivel de factor novedoso en los datos. El Capítulo 17 explora estos y otros métodos para codificar datos categóricos, más allá de simples variables ficticias o indicadoras.
Los diferentes pasos de una receta se comportan de manera diferente cuando se aplican a variables de los datos. Por ejemplo, step_log() modifica una columna en su lugar sin cambiar el nombre. Otros pasos, como step_dummy(), eliminan la columna de datos original y la reemplazan con una o más columnas con nombres diferentes. El efecto de un paso de receta depende del tipo de transformación de ingeniería de características que se realiza.
8.4.2 Términos de interacción
Los efectos de interacción involucran dos o más predictores. Tal efecto ocurre cuando un predictor tiene un efecto sobre el resultado que depende de uno o más predictores. Por ejemplo, si intenta predecir cuánto tráfico habrá durante su viaje, dos posibles predictores podrían ser la hora específica del día en que viaja y el clima. Sin embargo, la relación entre la cantidad de tráfico y el mal tiempo es diferente según el momento del día. En este caso, podría agregar un término de interacción entre los dos predictores al modelo junto con los dos predictores originales (que se denominan efectos principales). Numéricamente, un término de interacción entre predictores se codifica como su producto. Las interacciones se definen en términos de su efecto sobre el resultado y pueden ser combinaciones de diferentes tipos de datos (por ejemplo, numéricos, categóricos, etc.). Capítulo 7 de Kuhn y Johnson (2020) analiza las interacciones y cómo detectarlas con mayor detalle.
Después de explorar el conjunto de entrenamiento de Ames, podríamos encontrar que las pendientes de regresión para el área habitable bruta difieren para diferentes tipos de edificios, como se muestra en Figura 8.2.
ggplot(ames_train, aes(x = Gr_Liv_Area, y = 10^Sale_Price)) +
geom_point(alpha = .2) +
facet_wrap(~ Bldg_Type) +
geom_smooth(method = lm, formula = y ~ x, se = FALSE, color = "lightblue") +
scale_x_log10() +
scale_y_log10() +
labs(x = "Área Habitable Bruta", y = "Precio de Venta (USD)")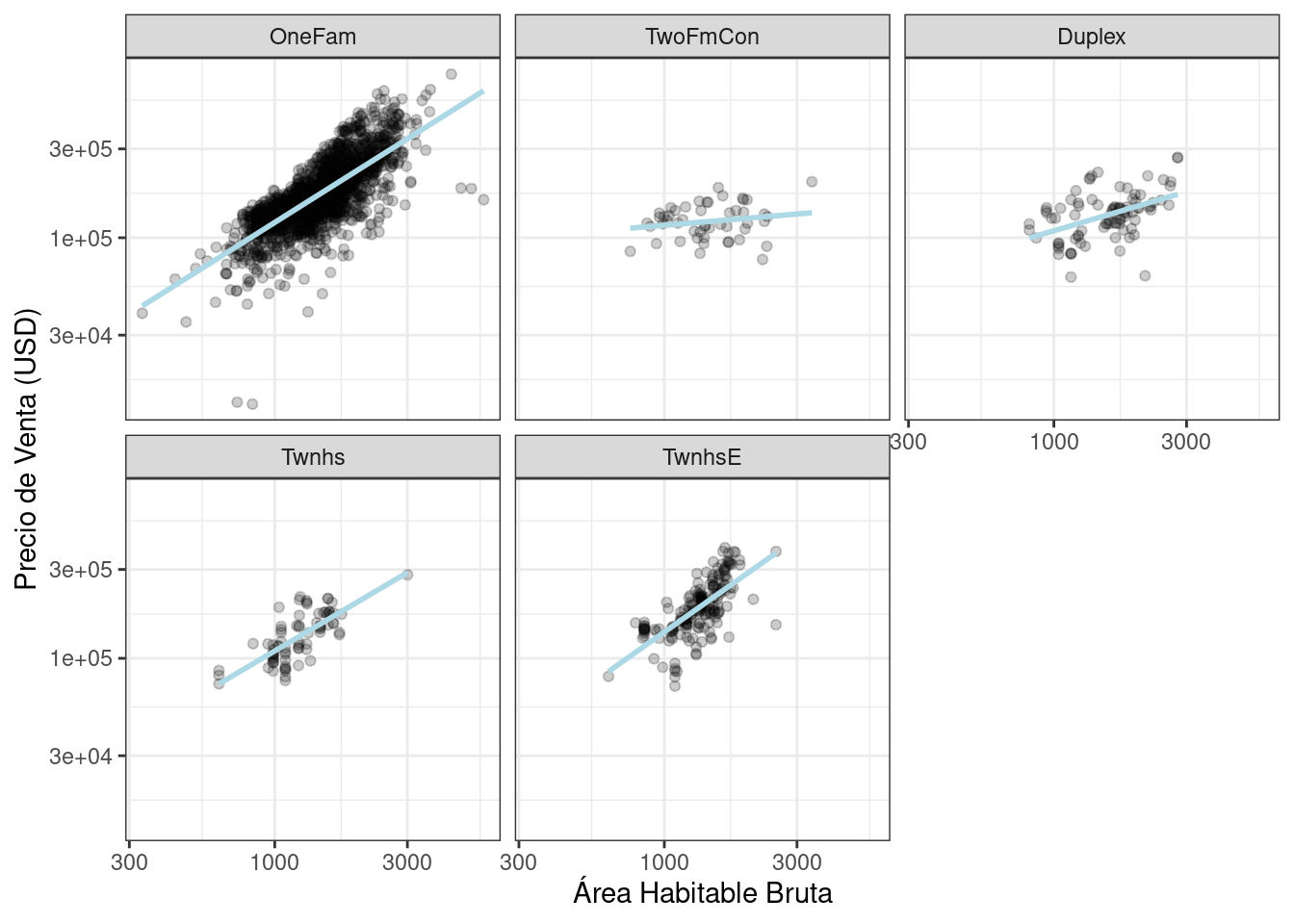
¿Cómo se especifican las interacciones en una receta? Una fórmula base R requeriría una interacción usando :, por lo que usaríamos:
Sale_Price ~ Neighborhood + log10(Gr_Liv_Area) + Bldg_Type +
log10(Gr_Liv_Area):Bldg_Type
# o
Sale_Price ~ Neighborhood + log10(Gr_Liv_Area) * Bldg_Type donde * expande esas columnas a los efectos principales y al término de interacción. Nuevamente, el método de fórmula hace muchas cosas simultáneamente y comprende que una variable de factor (como Bldg_Type) debe expandirse primero a variables ficticias y que la interacción debe involucrar a todas las columnas binarias resultantes.
Las recetas son más explícitas y secuenciales y te dan más control. Con la receta actual, step_dummy() ya ha creado variables ficticias. ¿Cómo los combinaríamos para una interacción? El paso adicional se vería así step_interact(~ términos de interacción) donde los términos en el lado derecho de la tilde son las interacciones. Estos pueden incluir selectores, por lo que sería apropiado utilizar:
simple_ames <-
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type,
data = ames_train) %>%
step_log(Gr_Liv_Area, base = 10) %>%
step_other(Neighborhood, threshold = 0.01) %>%
step_dummy(all_nominal_predictors()) %>%
# Gr_Liv_Area está en la escala logarítmica de un paso anterior
step_interact( ~ Gr_Liv_Area:starts_with("Bldg_Type_") )Se pueden especificar interacciones adicionales en esta fórmula separándolas por +. También tenga en cuenta que la receta sólo utilizará interacciones entre diferentes variables; si la fórmula usa var_1:var_1, este término se ignorará.
Supongamos que, en una receta, todavía no hemos creado variables ficticias para los tipos de edificios. Sería inapropiado incluir una columna de factores en este paso, como por ejemplo:
step_interact( ~ Gr_Liv_Area:Bldg_Type )Esto le dice al código subyacente (base R) utilizado por step_interact() para crear variables ficticias y luego formar las interacciones. De hecho, si esto ocurre, una advertencia indica que esto podría generar resultados inesperados.
Este comportamiento le brinda más control, pero es diferente de la fórmula del modelo estándar de R.
Al igual que con el nombre de variables ficticias, recipes proporciona nombres más coherentes para los términos de interacción. En este caso, la interacción se denomina Gr_Liv_Area_x_Bldg_Type_Duplex en lugar de Gr_Liv_Area:Bldg_TypeDuplex (que no es un nombre de columna válido para un marco de datos).
Recuerda que el orden importa. La superficie habitable bruta se transforma logarítmicamente antes del término de interacción. Las interacciones posteriores con esta variable también utilizarán la escala logarítmica.
8.4.3 Funciones splines
Cuando un predictor tiene una relación no lineal con el resultado, algunos tipos de modelos predictivos pueden aproximarse adaptativamente a esta relación durante el entrenamiento. Sin embargo, lo más simple suele ser mejor y no es raro intentar utilizar un modelo simple, como un ajuste lineal, y agregar características no lineales específicas para los predictores que puedan necesitarlas, como la longitud y la latitud para los datos de vivienda de Ames. Un método común para hacer esto es usar funciones spline para representar los datos. Los splines reemplazan el predictor numérico existente con un conjunto de columnas que permiten que un modelo emule una relación flexible y no lineal. A medida que se agregan más términos spline a los datos, aumenta la capacidad de representar la relación de forma no lineal. Desafortunadamente, también puede aumentar la probabilidad de detectar tendencias de datos que ocurren por casualidad (es decir, sobreajuste).
Si alguna vez usó geom_smooth() dentro de un ggplot, probablemente haya usado una representación spline de los datos. Por ejemplo, cada panel en Figura 8.3 utiliza un número diferente de splines suaves para el predictor de latitud:
library(patchwork)
library(splines)
plot_smoother <- function(deg_free) {
ggplot(ames_train, aes(x = Latitude, y = 10^Sale_Price)) +
geom_point(alpha = .2) +
scale_y_log10() +
geom_smooth(
method = lm,
formula = y ~ ns(x, df = deg_free),
color = "lightblue",
se = FALSE
) +
labs(title = paste(deg_free, "Términos Spline"),
y = "Precio de Venta (USD)")
}
( plot_smoother(2) + plot_smoother(5) ) / ( plot_smoother(20) + plot_smoother(100) )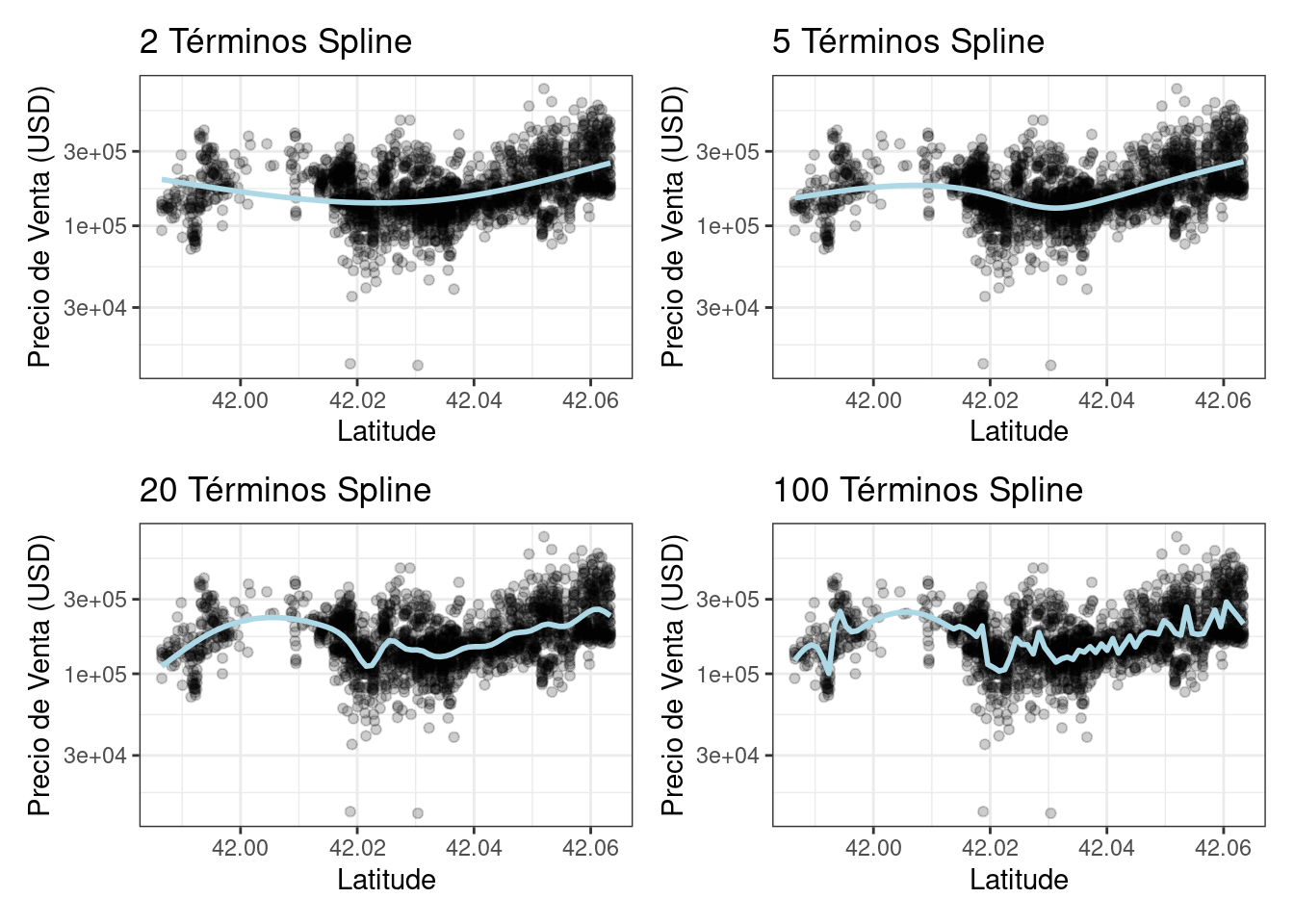
La función ns() en el paquete splines genera columnas de características usando funciones llamadas splines naturales.
Algunos paneles en Figura 8.3 claramente no encajan bien; dos términos no se ajustan bien a los datos, mientras que 100 términos se ajustan demasiado. Los paneles con veinticinco términos parecen ajustes razonablemente suaves que captan los patrones principales de los datos. Esto indica que la cantidad adecuada de “no linealidad” es importante. El número de términos spline podría entonces considerarse un parámetro de ajuste para este modelo. Estos tipos de parámetros se exploran en el Capítulo 12.
En recipes, varios pasos pueden crear este tipo de términos. Para agregar una representación spline natural para este predictor:
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type + Latitude,
data = ames_train) %>%
step_log(Gr_Liv_Area, base = 10) %>%
step_other(Neighborhood, threshold = 0.01) %>%
step_dummy(all_nominal_predictors()) %>%
step_interact( ~ Gr_Liv_Area:starts_with("Bldg_Type_") ) %>%
step_ns(Latitude, deg_free = 20)El usuario necesitaría determinar si tanto la vecindad como la latitud deberían estar en el modelo, ya que ambos representan los mismos datos subyacentes de diferentes maneras.
8.4.4 Extracción de características
Otro método común para representar múltiples características a la vez se llama extracción de características. La mayoría de estas técnicas crean nuevas características a partir de los predictores que capturan la información en un conjunto más amplio en su conjunto. Por ejemplo, el análisis de componentes principales (PCA) intenta extraer la mayor cantidad posible de información original en el conjunto de predictores utilizando un número menor de características. PCA es un método de extracción lineal, lo que significa que cada característica nueva es una combinación lineal de los predictores originales. Un aspecto interesante de PCA es que cada una de las nuevas características, llamadas componentes principales o puntuaciones de PCA, no están correlacionadas entre sí. Debido a esto, PCA puede resultar muy eficaz para reducir la correlación entre predictores. Tenga en cuenta que PCA sólo conoce los predictores; Es posible que las nuevas funciones de PCA no estén asociadas con el resultado.
En los datos de Ames, varios predictores miden el tamaño de la propiedad, como el tamaño total del sótano (Total_Bsmt_SF), el tamaño del primer piso (First_Flr_SF), la superficie habitable bruta (Gr_Liv_Area), etc. PCA podría ser una opción para representar estas variables potencialmente redundantes como un conjunto de características más pequeño. Además del área habitable bruta, estos predictores tienen el sufijo “SF” en sus nombres (para pies cuadrados), por lo que un paso de receta para PCA podría verse así:
# Utilice una expresión regular para capturar predictores del tamaño de la casa:
step_pca(matches("(SF$)|(Gr_Liv)"))Tenga en cuenta que todas estas columnas se miden en pies cuadrados. PCA supone que todos los predictores están en la misma escala. Eso es cierto en este caso, pero a menudo este paso puede ir precedido de step_normalize(), que centrará y escalará cada columna.
Existen pasos de recetas para otros métodos de extracción, como: análisis de componentes independientes (ICA), factorización matricial no negativa (NNMF), escalamiento multidimensional (MDS), aproximación y proyección de variedades uniformes (UMAP) y otros.
8.4.5 Pasos de muestreo de filas
Los pasos de una receta también pueden afectar las filas de un conjunto de datos. Por ejemplo, las técnicas de submuestreo para desequilibrios de clases cambian las proporciones de clases en los datos que se proporcionan al modelo; estas técnicas a menudo no mejoran el rendimiento general, pero pueden generar distribuciones de mejor comportamiento de las probabilidades de clase predichas. Estos son enfoques que puede probar al submuestrear sus datos con desequilibrio de clases:
Reducción de resolución los datos mantienen la clase minoritaria y toman una muestra aleatoria de la clase mayoritaria para que las frecuencias de las clases estén equilibradas.
Upsampling replica muestras de la clase minoritaria para equilibrar las clases. Algunas técnicas hacen esto sintetizando nuevas muestras que se asemejan a los datos de la clase minoritaria, mientras que otros métodos simplemente agregan las mismas muestras minoritarias repetidamente.
Los métodos híbridos hacen una combinación de ambos.
El paquete themis tiene pasos de receta que se pueden usar para abordar el desequilibrio de clases mediante submuestreo. Para una reducción de resolución simple, usaríamos:
step_downsample(outcome_column_name)Sólo el conjunto de entrenamiento debería verse afectado por estas técnicas. El conjunto de prueba u otras muestras reservadas deben dejarse como están cuando se procesan utilizando la receta. Por esta razón, todos los pasos de submuestreo tienen por defecto el argumento “skip” para que tenga un valor de “TRUE” (Sección 8.5).
Otras funciones de pasos también están basadas en filas: step_filter(), step_sample(), step_slice() y step_arrange(). En casi todos los usos de estos pasos, el argumento skip debe establecerse en TRUE.
8.4.6 Transformaciones generales
Reflejando la operación original dplyr, step_mutate() se puede utilizar para realizar una variedad de operaciones básicas con los datos. Se utiliza mejor para transformaciones sencillas como calcular una proporción de dos variables, como Bedroom_AbvGr / Full_Bath, la proporción entre dormitorios y baños para los datos de vivienda de Ames.
Al utilizar este paso flexible, tenga especial cuidado para evitar la fuga de datos en el preprocesamiento. Considere, por ejemplo, la transformación x = w > mean(w). Cuando se aplica a datos nuevos o datos de prueba, esta transformación usaría la media de “w” de los datos nuevos, no la media de “w” de los datos de entrenamiento.
8.4.7 Procesamiento natural del lenguaje
Las recetas también pueden manejar datos que no están en la estructura tradicional donde las columnas son características. Por ejemplo, el paquete textrecipes puede aplicar métodos de procesamiento de lenguaje natural a los datos. La columna de entrada suele ser una cadena de texto y se pueden utilizar diferentes pasos para tokenizar los datos (por ejemplo, dividir el texto en palabras separadas), filtrar tokens y crear nuevas características apropiadas para el modelado.
8.5 Saltarse Pasos Para Obtener Nuevos Datos
Los datos del precio de venta ya están transformados logarítmicamente en el marco de datos “ames”. ¿Por qué no utilizar?
step_log(Sale_Price, base = 10)Esto provocará un fallo cuando la receta se aplique a propiedades nuevas con un precio de venta desconocido. Dado que lo que intentamos predecir es el precio, probablemente no habrá una columna en los datos para esta variable. De hecho, para evitar la fuga de información, muchos paquetes de tidymodels aíslan los datos que se utilizan al realizar predicciones. Esto significa que el conjunto de entrenamiento y las columnas de resultados no están disponibles para su uso en el momento de la predicción.
Para transformaciones simples de la(s) columna(s) de resultados, sugerimos encarecidamente que esas operaciones se realicen fuera de la receta.
Sin embargo, hay otras circunstancias en las que esta no es una solución adecuada. Por ejemplo, en modelos de clasificación en los que existe un desequilibrio de clases grave, es común realizar un submuestreo de los datos que se proporcionan a la función de modelado. Por ejemplo, supongamos que hubiera dos clases y una tasa de eventos del 10 %. Un enfoque simple, aunque controvertido, sería reducir la muestra de los datos para que el modelo reciba todos los eventos y un 10% aleatorio de las muestras sin eventos.
El problema es que no se debe aplicar el mismo proceso de submuestreo a los datos que se predicen. Como resultado, cuando utilizamos una receta, necesitamos un mecanismo para garantizar que algunas operaciones se apliquen solo a los datos proporcionados al modelo. Cada función de paso tiene una opción llamada skip que, cuando se establece en TRUE, será ignorada por la función predict(). De esta manera, puede aislar los pasos que afectan los datos del modelado sin causar errores cuando se aplican a nuevas muestras. Sin embargo, todos los pasos se aplican cuando se usa fit().
Al momento de escribir este artículo, las funciones de paso en los paquetes recipes y themis que solo se aplican a los datos de entrenamiento son: step_adasyn(), step_bsmote(), step_downsample(), step_filter(), step_naomit(), step_nearmiss(), step_rose(), step_sample(), step_slice(), step_smote(), step_smotenc(), step_tomek(), and step_upsample().
8.6 Ordenar recipe()
En Sección 3.3, introdujimos el verbo tidy() para objetos estadísticos. También hay un método tidy() para recetas, así como pasos de recetas individuales. Antes de continuar, creemos una receta extendida para los datos de Ames usando algunos de los nuevos pasos que hemos discutido en este capítulo:
ames_rec <-
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type +
Latitude + Longitude, data = ames_train) %>%
step_log(Gr_Liv_Area, base = 10) %>%
step_other(Neighborhood, threshold = 0.01) %>%
step_dummy(all_nominal_predictors()) %>%
step_interact( ~ Gr_Liv_Area:starts_with("Bldg_Type_") ) %>%
step_ns(Latitude, Longitude, deg_free = 20)El método tidy(), cuando se llama con el objeto de receta, ofrece un resumen de los pasos de la receta:
tidy(ames_rec)
## # A tibble: 5 × 6
## number operation type trained skip id
## <int> <chr> <chr> <lgl> <lgl> <chr>
## 1 1 step log FALSE FALSE log_66JTU
## 2 2 step other FALSE FALSE other_ePfcw
## 3 3 step dummy FALSE FALSE dummy_Z18Cl
## 4 4 step interact FALSE FALSE interact_JLU36
## 5 5 step ns FALSE FALSE ns_rvsqQEste resultado puede ser útil para identificar pasos individuales, quizás para luego poder ejecutar el método tidy() en un paso específico.
Podemos especificar el argumento id en cualquier llamada de función de paso; de lo contrario, se genera utilizando un sufijo aleatorio. Establecer este valor puede resultar útil si se agrega el mismo tipo de paso a la receta más de una vez. Especifiquemos el id con anticipación para step_other(), ya que queremos tidy():
ames_rec <-
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type +
Latitude + Longitude, data = ames_train) %>%
step_log(Gr_Liv_Area, base = 10) %>%
step_other(Neighborhood, threshold = 0.01, id = "my_id") %>%
step_dummy(all_nominal_predictors()) %>%
step_interact( ~ Gr_Liv_Area:starts_with("Bldg_Type_") ) %>%
step_ns(Latitude, Longitude, deg_free = 20)Reacondicionaremos el flujo de trabajo con esta nueva receta:
lm_wflow <-
workflow() %>%
add_model(lm_model) %>%
add_recipe(ames_rec)
lm_fit <- fit(lm_wflow, ames_train)El método tidy() se puede volver a llamar junto con el identificador id que especificamos para obtener nuestros resultados al aplicar step_other():
estimated_recipe <-
lm_fit %>%
extract_recipe(estimated = TRUE)
tidy(estimated_recipe, id = "my_id")
## # A tibble: 22 × 3
## terms retained id
## <chr> <chr> <chr>
## 1 Neighborhood North_Ames my_id
## 2 Neighborhood College_Creek my_id
## 3 Neighborhood Old_Town my_id
## 4 Neighborhood Edwards my_id
## 5 Neighborhood Somerset my_id
## 6 Neighborhood Northridge_Heights my_id
## # ℹ 16 more rowsLos resultados de tidy() que vemos aquí al usar step_other() muestran qué niveles de factor se retuvieron, es decir, no se agregaron a la nueva categoría “otros”.
El método tidy() también se puede llamar con el identificador number, si sabemos qué paso de la receta necesitamos:
tidy(estimated_recipe, number = 2)
## # A tibble: 22 × 3
## terms retained id
## <chr> <chr> <chr>
## 1 Neighborhood North_Ames my_id
## 2 Neighborhood College_Creek my_id
## 3 Neighborhood Old_Town my_id
## 4 Neighborhood Edwards my_id
## 5 Neighborhood Somerset my_id
## 6 Neighborhood Northridge_Heights my_id
## # ℹ 16 more rowsCada método tidy() devuelve la información relevante sobre ese paso. Por ejemplo, el método tidy() para step_dummy() devuelve una columna con las variables que se convirtieron en variables ficticias y otra columna con todos los niveles conocidos para cada columna.
8.7 Roles De Columna
Cuando se usa una fórmula con la llamada inicial a receta(), asigna roles a cada una de las columnas, dependiendo de en qué lado de la tilde se encuentran. Esos roles son "predictor" o "outcome". Sin embargo, se pueden asignar otros roles según sea necesario.
Por ejemplo, en nuestro conjunto de datos de Ames, los datos originales sin procesar contenían una columna para la dirección.1 Puede resultar útil mantener esa columna en los datos para que, después de realizar las predicciones, se obtengan resultados problemáticos. se puede investigar en detalle. En otras palabras, la columna podría ser importante incluso cuando no sea un predictor o un resultado.
Para resolver esto, las funciones add_role(), remove_role() y update_role() pueden resultar útiles. Por ejemplo, para los datos del precio de la vivienda, la función de la columna de dirección postal podría modificarse usando:
ames_rec %>% update_role(address, new_role = "street address")Después de este cambio, la columna address en el marco de datos ya no será un predictor sino una street address de acuerdo con la receta. Se puede utilizar cualquier cadena de caracteres como rol. Además, las columnas pueden tener múltiples roles (se agregan roles adicionales mediante add_role()) para que puedan seleccionarse en más de un contexto.
Esto puede resultar útil cuando los datos se vuelven a muestrear. Ayuda a mantener las columnas que no están involucradas con el modelo encajadas en el mismo marco de datos (en lugar de en un vector externo). El remuestreo, descrito en el Capítulo 10, crea versiones alternativas de los datos principalmente mediante submuestreo de filas. Si la dirección postal estuviera en otra columna, se requeriría un submuestreo adicional y podría generar un código más complejo y una mayor probabilidad de errores.
Finalmente, todas las funciones de paso tienen un campo “rol” que puede asignar roles a los resultados del paso. En muchos casos, las columnas afectadas por un paso conservan su función actual. Por ejemplo, las llamadas step_log() a nuestro objeto ames_rec afectaron la columna Gr_Liv_Area. Para ese paso, el comportamiento predeterminado es mantener la función existente para esta columna, ya que no se crea ninguna columna nueva. Como contraejemplo, el paso para producir splines establece de forma predeterminada que las nuevas columnas tengan una función de "predictor" ya que así es como normalmente se usan las columnas spline en un modelo. La mayoría de los pasos tienen valores predeterminados razonables pero, dado que los valores predeterminados pueden ser diferentes, asegúrese de consultar la página de documentación para comprender qué roles se asignarán.
8.8 Resumen Del Capítulo
En este capítulo, aprendió a usar recipes para ingeniería de funciones flexible y preprocesamiento de datos, desde la creación de variables ficticias hasta el manejo del desequilibrio de clases y más. La ingeniería de características es una parte importante del proceso de modelado donde puede ocurrir fácilmente una fuga de información y se deben adoptar buenas prácticas. Entre el paquete recipes y otros paquetes que amplían recetas, hay más de 100 pasos disponibles. Todos los pasos posibles de la receta se enumeran en tidymodels.org/find. El marco recipes proporciona un rico entorno de manipulación de datos para preprocesar y transformar datos antes del modelado. Además, tidymodels.org/learn/develop/recipes/ muestra cómo se pueden crear pasos personalizados.
Nuestro trabajo aquí ha utilizado recetas únicamente dentro de un objeto de flujo de trabajo. Para el modelado, ese es el uso recomendado porque la ingeniería de características debe estimarse junto con un modelo. Sin embargo, para la visualización y otras actividades, un flujo de trabajo puede no ser apropiado; Es posible que se requieran funciones más específicas de recetas. El Capítulo 16 analiza las API de nivel inferior para adaptar, usar y solucionar problemas de recetas.
El código que usaremos en capítulos posteriores es:
library(tidymodels)
data(ames)
ames <- mutate(ames, Sale_Price = log10(Sale_Price))
set.seed(502)
ames_split <- initial_split(ames, prop = 0.80, strata = Sale_Price)
ames_train <- training(ames_split)
ames_test <- testing(ames_split)
ames_rec <-
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type +
Latitude + Longitude, data = ames_train) %>%
step_log(Gr_Liv_Area, base = 10) %>%
step_other(Neighborhood, threshold = 0.01) %>%
step_dummy(all_nominal_predictors()) %>%
step_interact( ~ Gr_Liv_Area:starts_with("Bldg_Type_") ) %>%
step_ns(Latitude, Longitude, deg_free = 20)
lm_model <- linear_reg() %>% set_engine("lm")
lm_wflow <-
workflow() %>%
add_model(lm_model) %>%
add_recipe(ames_rec)
lm_fit <- fit(lm_wflow, ames_train)Nuestra versión de estos datos no contiene esa columna.↩︎