3 Vectores
3.1 Introducción
Este capítulo analiza la familia más importante de tipos de datos en base R: vectores1. Si bien es probable que ya haya usado muchos (si no todos) de los diferentes tipos de vectores, es posible que no haya pensado profundamente en cómo están interrelacionados. En este capítulo, no cubriré los tipos de vectores individuales con demasiado detalle, pero le mostraré cómo encajan todos los tipos como un todo. Si necesita más detalles, puede encontrarlos en la documentación de R.
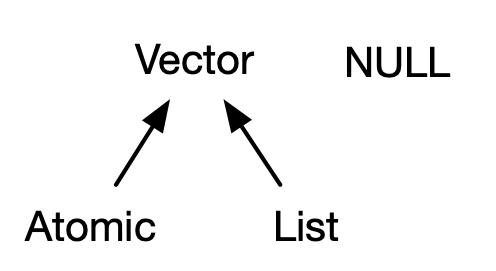
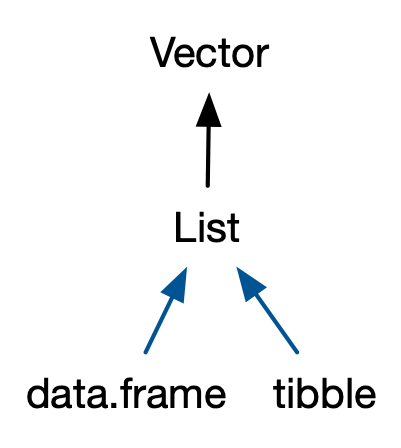
Los vectores vienen en dos sabores: vectores atómicos y listas 2. Se diferencian en cuanto a los tipos de sus elementos: para los vectores atómicos, todos los elementos deben tener el mismo tipo; para las listas, los elementos pueden tener diferentes tipos. Si bien no es un vector, NULL está estrechamente relacionado con los vectores y, a menudo, cumple la función de un vector genérico de longitud cero. Este diagrama, que ampliaremos a lo largo de este capítulo, ilustra las relaciones básicas:
Cada vector también puede tener atributos, que puede considerar como una lista con nombre de metadatos arbitrarios. Dos atributos son particularmente importantes. El atributo dimensión convierte los vectores en matrices y arreglos y el atributo clase impulsa el sistema de objetos S3. Si bien aprenderá a usar S3 en el Capítulo 13, aquí aprenderá sobre algunos de los vectores S3 más importantes: factores, fecha y hora, data frames y tibbles. Y aunque las estructuras 2D como matrices y data frames no son necesariamente lo que le viene a la mente cuando piensa en vectores, también aprenderá por qué R los considera vectores.
Prueba
Responda este breve cuestionario para determinar si necesita leer este capítulo. Si las respuestas le vienen a la mente rápidamente, puede saltarse cómodamente este capítulo. Puede comprobar sus respuestas en la Sección 3.8.
¿Cuáles son los cuatro tipos comunes de vectores atómicos? ¿Cuáles son los dos tipos raros?
¿Qué son los atributos? ¿Cómo los consigues y los configuras?
¿En qué se diferencia una lista de un vector atómico? ¿En qué se diferencia una matriz de un data frame?
¿Puedes tener una lista que sea una matriz? ¿Puede un data frame tener una columna que sea una matriz?
¿En qué se diferencian los tibbles de los data frames?
Estructura
La Sección 3.2 te introduce a los vectores atómicos: lógico, entero, doble y de carácter. Estas son las estructuras de datos más simples de R.
La Sección 3.3 toma un pequeño desvío para discutir los atributos, la especificación de metadatos flexibles de R. Los atributos más importantes son los nombres, las dimensiones y la clase.
La Sección 3.4 analiza los tipos de vectores importantes que se construyen combinando vectores atómicos con atributos especiales. Estos incluyen factores, fechas, fechas-horas y duraciones.
La Sección 3.5 se sumerge en las listas. Las listas son muy similares a los vectores atómicos, pero tienen una diferencia clave: un elemento de una lista puede ser cualquier tipo de datos, incluida otra lista. Esto los hace adecuados para representar datos jerárquicos.
La Sección 3.6 te enseña sobre data frames y tibbles, que se utilizan para representar datos rectangulares. Combinan el comportamiento de listas y matrices para crear una estructura ideal para las necesidades de los datos estadísticos.
3.2 Vectores atómicos
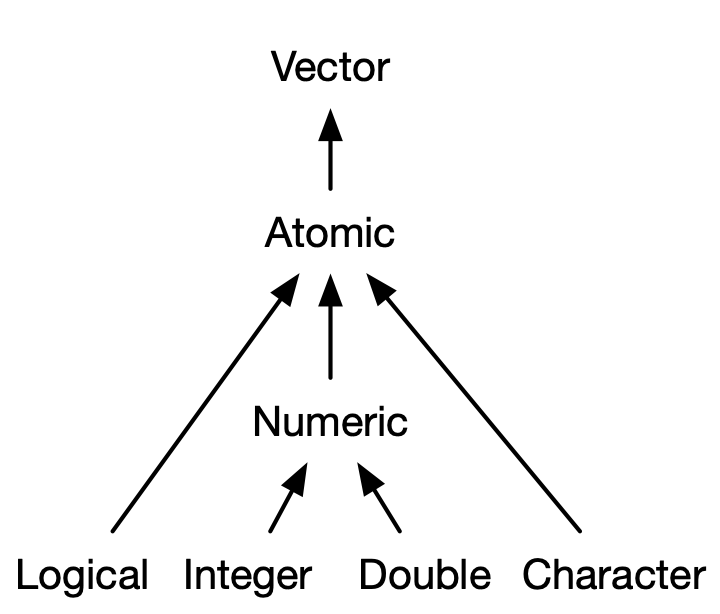
Hay cuatro tipos principales de vectores atómicos: lógico, entero, doble y carácter (que contiene cadenas). En conjunto, los vectores enteros y dobles se conocen como vectores numéricos 3. Hay dos tipos raros: complejos y crudos. No los discutiré más porque los números complejos rara vez se necesitan en las estadísticas, y los vectores sin procesar son un tipo especial que solo se necesita cuando se manejan datos binarios.
3.2.1 Escalares
Cada uno de los cuatro tipos principales tiene una sintaxis especial para crear un valor individual, también conocido como escalar4:
Los lógicos se pueden escribir completos (
TRUEoFALSE) o abreviados (ToF).Los dobles se pueden especificar en formato decimal (
0.1234), científico (1.23e4) o hexadecimal (0xcafe). Hay tres valores especiales únicos para los dobles:Inf,-InfyNaN(no es un número). Estos son valores especiales definidos por el estándar de punto flotante.Los enteros se escriben de forma similar a los dobles, pero deben ir seguidos de
L5 (1234L,1e4Lo0xcafeL), y no pueden contener valores fraccionarios.Las cadenas están rodeadas por
"("hola") o'('adiós'). Los caracteres especiales se escapan con\; consulte?Quotespara obtener detalles completos.
3.2.2 Crear vectores más largos con c()
Para crear vectores más largos a partir de otros más cortos, use c(), abreviatura de combinar:
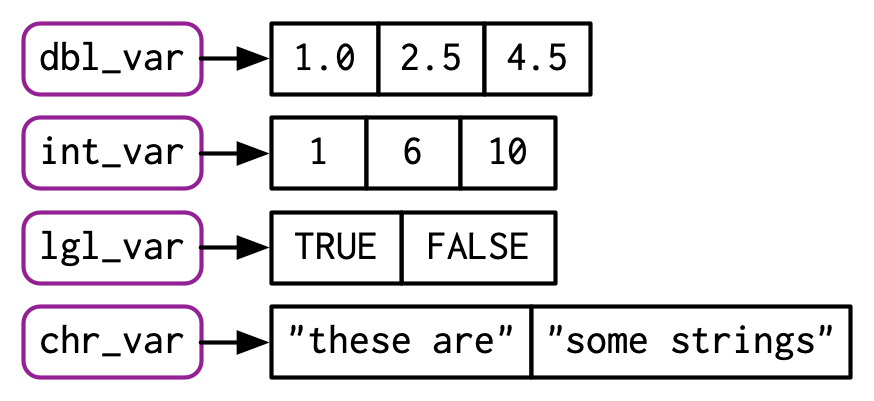
lgl_var <- c(TRUE, FALSE)
int_var <- c(1L, 6L, 10L)
dbl_var <- c(1, 2.5, 4.5)
chr_var <- c("these are", "some strings")Cuando las entradas son vectores atómicos, c() siempre crea otro vector atómico; es decir, se aplana:
c(c(1, 2), c(3, 4))
#> [1] 1 2 3 4En los diagramas, representaré los vectores como rectángulos conectados, por lo que el código anterior podría dibujarse de la siguiente manera:
Puedes determinar el tipo de un vector con typeof()6 y su longitud con length().
typeof(lgl_var)
#> [1] "logical"
typeof(int_var)
#> [1] "integer"
typeof(dbl_var)
#> [1] "double"
typeof(chr_var)
#> [1] "character"3.2.3 Valores Faltantes
R representa valores faltantes o desconocidos, con un valor centinela especial: NA (abreviatura de no aplicable). Los valores faltantes tienden a ser infecciosos: la mayoría de los cálculos que involucran un valor faltante devolverán otro valor faltante.
NA > 5
#> [1] NA
10 * NA
#> [1] NA
!NA
#> [1] NASólo hay unas pocas excepciones a esta regla. Estos ocurren cuando alguna identidad se mantiene para todas las entradas posibles:
NA ^ 0
#> [1] 1
NA | TRUE
#> [1] TRUE
NA & FALSE
#> [1] FALSELa propagación de faltantes conduce a un error común al determinar qué valores faltan en un vector:
x <- c(NA, 5, NA, 10)
x == NA
#> [1] NA NA NA NAEste resultado es correcto (aunque un poco sorprendente) porque no hay motivo para creer que un valor faltante tiene el mismo valor que otro. En su lugar, use is.na() para probar la presencia de ausencias:
is.na(x)
#> [1] TRUE FALSE TRUE FALSENB: Técnicamente, hay cuatro valores faltantes7, uno para cada uno de los tipos atómicos: NA (lógico), NA_integer_ (entero), NA_real_ (doble) y NA_character_ (carácter). Esta distinción generalmente no es importante porque ‘NA’ será forzado automáticamente al tipo correcto cuando sea necesario.
3.2.4 Pruebas y coerción
En general, puede probar si un vector es de un tipo dado con una función is.*(), pero estas funciones deben usarse con cuidado. is.logical(), is.integer(), is.double() y is.character() hacen lo que cabría esperar: prueban si un vector es un carácter, doble, entero o lógico. Evite is.vector(), is.atomic() y is.numeric(): no comprueban si tiene un vector, un vector atómico o un vector numérico; deberá leer detenidamente la documentación para descubrir qué es lo que realmente hacen.
Para los vectores atómicos, el tipo es una propiedad de todo el vector: todos los elementos deben ser del mismo tipo. Cuando intente combinar diferentes tipos, se coaccionarán en un orden fijo: carácter → doble → entero → lógico. Por ejemplo, la combinación de un carácter y un número entero produce un carácter:
str(c("a", 1))
#> chr [1:2] "a" "1"La coerción a menudo ocurre automáticamente. La mayoría de las funciones matemáticas (+, log, abs, etc.) se convertirán en numéricas. Esta coerción es particularmente útil para vectores lógicos porque TRUE se convierte en 1 y FALSE se convierte en 0.
x <- c(FALSE, FALSE, TRUE)
as.numeric(x)
#> [1] 0 0 1
# Número total de VERDADEROS
sum(x)
#> [1] 1
# Proporción que son VERDADERAS
mean(x)
#> [1] 0.333En general, puede forzar deliberadamente usando una función as.*(), como as.logical(), as.integer(), as.double() o as.character(). La coerción fallida de cadenas genera una advertencia y un valor faltante:
as.integer(c("1", "1.5", "a"))
#> Warning: NAs introduced by coercion
#> [1] 1 1 NA3.2.5 Ejercicios
¿Cómo se crean escalares crudos y complejos? (Ver
?rawy?complex.)Pon a prueba tu conocimiento de las reglas de coerción de vectores prediciendo el resultado de los siguientes usos de
c():c(1, FALSE) c("a", 1) c(TRUE, 1L)¿Por qué
1 == "1"es verdadero? ¿Por qué-1 < FALSEes verdadero? ¿Por qué"one" < 2es falso?¿Por qué el valor faltante predeterminado,
NA, es un vector lógico? ¿Qué tienen de especial los vectores lógicos? (Pista: piensa enc(FALSE, NA_character_).)Precisamente, ¿qué prueban
is.atomic(),is.numeric()yis.vector()?
3.3 Atributos
Es posible que haya notado que el conjunto de vectores atómicos no incluye una serie de estructuras de datos importantes como matrices, arreglos, factores o fechas y horas. Estos tipos se construyen sobre vectores atómicos agregando atributos. En esta sección, aprenderá los conceptos básicos de los atributos y cómo el atributo dim crea matrices y arreglos. En la siguiente sección, aprenderá cómo se usa el atributo de clase para crear vectores de S3, incluidos factores, fechas y fechas y horas.
3.3.1 Conseguir y configurar
Puede pensar en los atributos como pares de nombre-valor 8 que adjuntan metadatos a un objeto. Los atributos individuales pueden recuperarse y modificarse con attr(), o recuperarse en masa con attributes(), y establecerse en masa con structure().
a <- 1:3
attr(a, "x") <- "abcdef"
attr(a, "x")
#> [1] "abcdef"
attr(a, "y") <- 4:6
str(attributes(a))
#> List of 2
#> $ x: chr "abcdef"
#> $ y: int [1:3] 4 5 6
# Or equivalently
a <- structure(
1:3,
x = "abcdef",
y = 4:6
)
str(attributes(a))
#> List of 2
#> $ x: chr "abcdef"
#> $ y: int [1:3] 4 5 6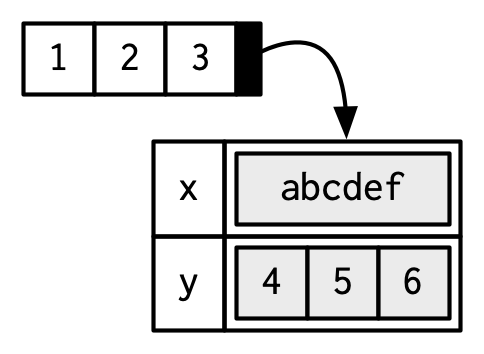
En general, los atributos deben considerarse efímeros. Por ejemplo, la mayoría de los atributos se pierden en la mayoría de las operaciones:
attributes(a[1])
#> NULL
attributes(sum(a))
#> NULLSolo hay dos atributos que se conservan de forma rutinaria:
- names, un vector de caracteres que da a cada elemento un nombre.
- dim, abreviatura de dimensiones, un vector entero, que se utiliza para convertir vectores en matrices o arreglos.
Para conservar otros atributos, deberá crear su propia clase S3, el tema del Capítulo 13.
3.3.2 Nombres
Puede nombrar un vector de tres maneras:
# Cuando lo crea:
x <- c(a = 1, b = 2, c = 3)
# Al asignar un vector de caracteres a names()
x <- 1:3
names(x) <- c("a", "b", "c")
# En línea con setNames():
x <- setNames(1:3, c("a", "b", "c"))Evite usar attr(x, "names") ya que requiere escribir más y es menos legible que names(x). Puede eliminar nombres de un vector usando x <- unname(x) o names(x) <- NULL.
Para ser técnicamente correcto, al dibujar el vector nombrado x, debería dibujarlo así:
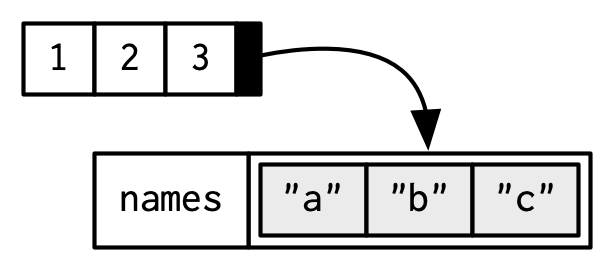
Sin embargo, los nombres son tan especiales e importantes que, a menos que intente llamar la atención específicamente sobre la estructura de datos de los atributos, los usaré para etiquetar el vector directamente:
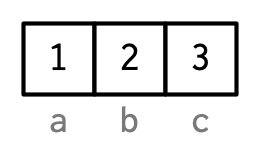
Para que sea útil con subconjuntos de caracteres (p. ej., la Sección 4.5.1), los nombres deben ser únicos y no faltantes, pero R no impone esto. Dependiendo de cómo se establezcan los nombres, los nombres faltantes pueden ser "" o NA_character_. Si faltan todos los nombres, names() devolverá NULL.
3.3.3 Dimensiones
Agregar un atributo dim a un vector le permite comportarse como una matriz bidimensional o una matriz multidimensional. Las matrices y los arreglos son principalmente herramientas matemáticas y estadísticas, no herramientas de programación, por lo que se usarán con poca frecuencia y solo se tratarán brevemente en este libro. Su característica más importante es el subconjunto multidimensional, que se trata en la Sección 4.2.3.
Puede crear matrices y arreglos con matrix() y array(), o usando el formulario de asignación de dim():
# Dos argumentos escalares especifican tamaños de fila y columna
x <- matrix(1:6, nrow = 2, ncol = 3)
x
#> [,1] [,2] [,3]
#> [1,] 1 3 5
#> [2,] 2 4 6
# Un argumento vectorial para describir todas las dimensiones
y <- array(1:12, c(2, 3, 2))
y
#> , , 1
#>
#> [,1] [,2] [,3]
#> [1,] 1 3 5
#> [2,] 2 4 6
#>
#> , , 2
#>
#> [,1] [,2] [,3]
#> [1,] 7 9 11
#> [2,] 8 10 12
# También puede modificar un objeto en su lugar configurando dim()
z <- 1:6
dim(z) <- c(3, 2)
z
#> [,1] [,2]
#> [1,] 1 4
#> [2,] 2 5
#> [3,] 3 6Muchas de las funciones para trabajar con vectores tienen generalizaciones para matrices y arreglos:
| Vector | Matriz | Arreglo |
|---|---|---|
names() |
rownames(), colnames() |
dimnames() |
length() |
nrow(), ncol() |
dim() |
c() |
rbind(), cbind() |
abind::abind() |
| — | t() |
aperm() |
is.null(dim(x)) |
is.matrix() |
is.array() |
Un vector sin un conjunto de atributos “dim” a menudo se considera unidimensional, pero en realidad tiene dimensiones NULL. También puede tener matrices con una sola fila o una sola columna, o arreglos con una sola dimensión. Pueden imprimir de manera similar, pero se comportarán de manera diferente. Las diferencias no son demasiado importantes, pero es útil saber que existen en caso de que obtenga un resultado extraño de una función (tapply() es un infractor frecuente). Como siempre, usa str() para revelar las diferencias.
str(1:3) # 1d vector
#> int [1:3] 1 2 3
str(matrix(1:3, ncol = 1)) # column vector
#> int [1:3, 1] 1 2 3
str(matrix(1:3, nrow = 1)) # row vector
#> int [1, 1:3] 1 2 3
str(array(1:3, 3)) # "array" vector
#> int [1:3(1d)] 1 2 33.3.4 Ejercicios
¿Cómo se implementa
setNames()? ¿Cómo se implementaunname()? Lee el código fuente.¿Qué devuelve
dim()cuando se aplica a un vector unidimensional? ¿Cuándo podría usarNROW()oNCOL()?¿Cómo describirías los siguientes tres objetos? ¿Qué los hace diferentes de
1:5?x1 <- array(1:5, c(1, 1, 5)) x2 <- array(1:5, c(1, 5, 1)) x3 <- array(1:5, c(5, 1, 1))Un borrador inicial usó este código para ilustrar
structure():structure(1:5, comment = "my attribute") #> [1] 1 2 3 4 5Pero cuando imprime ese objeto, no ve el atributo de comentario. ¿Por qué? ¿Falta el atributo o hay algo más especial en él? (Sugerencia: intente usar la ayuda).
3.4 Vectores atómicos S3
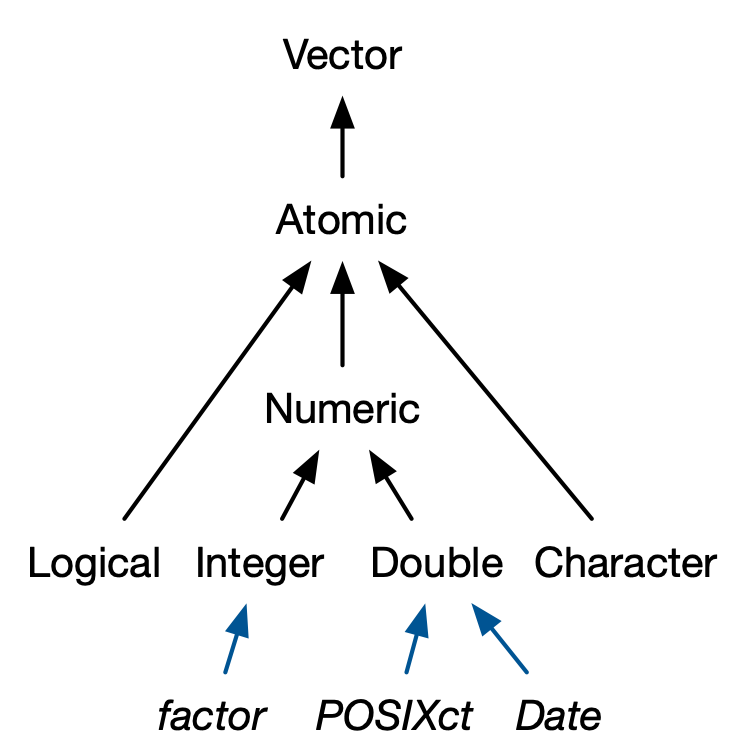
Uno de los atributos vectoriales más importantes es la clase, que subyace en el sistema de objetos S3. Tener un atributo de clase convierte un objeto en un objeto S3, lo que significa que se comportará de manera diferente a un vector regular cuando se pasa a una función genérica. Cada objeto de S3 se crea sobre un tipo base y, a menudo, almacena información adicional en otros atributos. Aprenderá los detalles del sistema de objetos de S3 y cómo crear sus propias clases de S3 en el Capítulo 13.
En esta sección, analizaremos cuatro vectores S3 importantes que se utilizan en la base R:
Datos categóricos, donde los valores provienen de un conjunto fijo de niveles registrados en vectores de factores.
Fechas (con resolución de día), que se registran en vectores Fecha.
Fechas-horas (con resolución de segundos o subsegundos), que se almacenan en vectores POSIXct.
Duraciones, que se almacenan en vectores difftime.
3.4.1 Factores
Un factor es un vector que solo puede contener valores predefinidos. Se utiliza para almacenar datos categóricos. Los factores se construyen sobre un vector entero con dos atributos: una class, “factor”, que hace que se comporte de manera diferente a los vectores enteros normales, y levels, que define el conjunto de valores permitidos.
x <- factor(c("a", "b", "b", "a"))
x
#> [1] a b b a
#> Levels: a b
typeof(x)
#> [1] "integer"
attributes(x)
#> $levels
#> [1] "a" "b"
#>
#> $class
#> [1] "factor"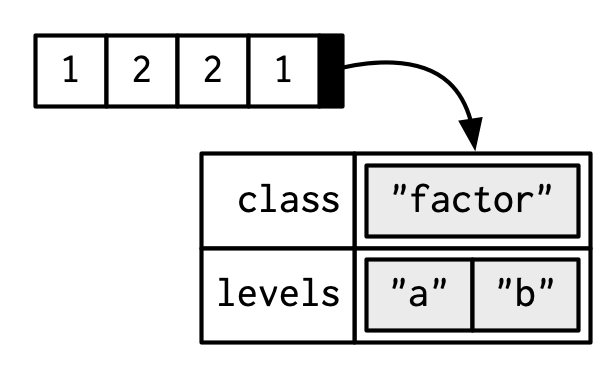
Los factores son útiles cuando conoce el conjunto de valores posibles, pero no todos están presentes en un conjunto de datos determinado. A diferencia de un vector de caracteres, cuando tabula un factor obtendrá recuentos de todas las categorías, incluso las no observadas:
sex_char <- c("m", "m", "m")
sex_factor <- factor(sex_char, levels = c("m", "f"))
table(sex_char)
#> sex_char
#> m
#> 3
table(sex_factor)
#> sex_factor
#> m f
#> 3 0Los factores ordenados son una variación menor de los factores. En general, se comportan como factores regulares, pero el orden de los niveles es significativo (bajo, medio, alto) (una propiedad que algunas funciones de modelado y visualización aprovechan automáticamente).
grade <- ordered(c("b", "b", "a", "c"), levels = c("c", "b", "a"))
grade
#> [1] b b a c
#> Levels: c < b < aEn base R9, tiende a encontrar factores con mucha frecuencia porque muchas funciones de base R (como read.csv() y data.frame()) convierten automáticamente los vectores de caracteres en factores. Esto es subóptimo porque no hay forma de que esas funciones conozcan el conjunto de todos los niveles posibles o su orden correcto: los niveles son una propiedad de la teoría o el diseño experimental, no de los datos. En su lugar, utilice el argumento stringsAsFactors = FALSE para suprimir este comportamiento y luego convierta manualmente los vectores de caracteres en factores usando su conocimiento de los datos “teóricos”. Para conocer el contexto histórico de este comportamiento, recomiendo stringsAsFactors: Una biografía no autorizada de Roger Peng, y stringsAsFactors = <sigh> de Thomas Lumley.
Si bien los factores se ven (y a menudo se comportan como) vectores de caracteres, se construyen sobre números enteros. Así que tenga cuidado al tratarlos como cadenas. Algunos métodos de cadena (como gsub() y grepl()) forzarán automáticamente los factores a cadenas, otros (como nchar()) generarán un error y otros (como c()) usarán los valores enteros subyacentes. Por esta razón, normalmente es mejor convertir explícitamente los factores en vectores de caracteres si necesita un comportamiento similar al de una cadena.
3.4.2 Fechas
Los vectores de fecha se construyen sobre vectores dobles. Tienen clase “Date” y ningún otro atributo:
today <- Sys.Date()
typeof(today)
#> [1] "double"
attributes(today)
#> $class
#> [1] "Date"El valor del doble (que se puede ver quitando la clase), representa el número de días desde 1970-01-0110:
date <- as.Date("1970-02-01")
unclass(date)
#> [1] 313.4.3 Fecha-Hora
Base R11 proporciona dos formas de almacenar información de fecha y hora, POSIXct y POSIXlt. Estos son nombres ciertamente extraños: “POSIX” es la abreviatura de Portable Operating System Interface, que es una familia de estándares multiplataforma. “ct” representa la hora del calendario (el tipo time_t en C), y “lt” la hora local (el tipo struct tm en C). Aquí nos centraremos en POSIXct, porque es el más simple, está construido sobre un vector atómico y es más apropiado para usar en tramas de datos. Los vectores POSIXct se construyen sobre vectores dobles, donde el valor representa la cantidad de segundos desde 1970-01-01.
now_ct <- as.POSIXct("2018-08-01 22:00", tz = "UTC")
now_ct
#> [1] "2018-08-01 22:00:00 UTC"
typeof(now_ct)
#> [1] "double"
attributes(now_ct)
#> $class
#> [1] "POSIXct" "POSIXt"
#>
#> $tzone
#> [1] "UTC"El atributo tzone controla solo cómo se formatea la fecha y la hora; no controla el instante de tiempo representado por el vector. Tenga en cuenta que la hora no se imprime si es medianoche.
structure(now_ct, tzone = "Asia/Tokyo")
#> [1] "2018-08-02 07:00:00 JST"
structure(now_ct, tzone = "America/New_York")
#> [1] "2018-08-01 18:00:00 EDT"
structure(now_ct, tzone = "Australia/Lord_Howe")
#> [1] "2018-08-02 08:30:00 +1030"
structure(now_ct, tzone = "Europe/Paris")
#> [1] "2018-08-02 CEST"3.4.4 Duraciones
Las duraciones, que representan la cantidad de tiempo entre pares de fechas o fechas-horas, se almacenan en difftimes. Los tiempos de diferencia se construyen sobre los dobles y tienen un atributo de unidades que determina cómo se debe interpretar el número entero:
one_week_1 <- as.difftime(1, units = "weeks")
one_week_1
#> Time difference of 1 weeks
typeof(one_week_1)
#> [1] "double"
attributes(one_week_1)
#> $class
#> [1] "difftime"
#>
#> $units
#> [1] "weeks"
one_week_2 <- as.difftime(7, units = "days")
one_week_2
#> Time difference of 7 days
typeof(one_week_2)
#> [1] "double"
attributes(one_week_2)
#> $class
#> [1] "difftime"
#>
#> $units
#> [1] "days"3.4.5 Ejercicios
¿Qué tipo de objeto devuelve
table()? ¿Cuál es su tipo? ¿Qué atributos tiene? ¿Cómo cambia la dimensionalidad a medida que tabula más variables?¿Qué le sucede a un factor cuando modificas sus niveles?
f1 <- factor(letters) levels(f1) <- rev(levels(f1))¿Qué hace este código? ¿En qué se diferencian
f2yf3def1?f2 <- rev(factor(letters)) f3 <- factor(letters, levels = rev(letters))
3.5 Listas
Las listas son un paso más en complejidad que los vectores atómicos: cada elemento puede ser de cualquier tipo, no solo vectores. Técnicamente hablando, cada elemento de una lista es en realidad del mismo tipo porque, como viste en la Sección 2.3.3, cada elemento es realmente una referencia a otro objeto, que puede ser de cualquier tipo.
3.5.1 Creando
Construyes listas con list():
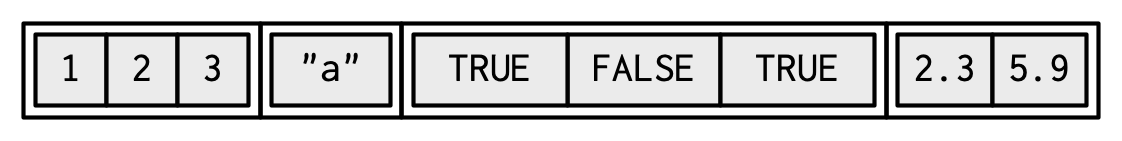
l1 <- list(
1:3,
"a",
c(TRUE, FALSE, TRUE),
c(2.3, 5.9)
)
typeof(l1)
#> [1] "list"
str(l1)
#> List of 4
#> $ : int [1:3] 1 2 3
#> $ : chr "a"
#> $ : logi [1:3] TRUE FALSE TRUE
#> $ : num [1:2] 2.3 5.9Dado que los elementos de una lista son referencias, crear una lista no implica copiar los componentes en la lista. Por esta razón, el tamaño total de una lista puede ser más pequeño de lo esperado.
lobstr::obj_size(mtcars)
#> 7.21 kB
l2 <- list(mtcars, mtcars, mtcars, mtcars)
lobstr::obj_size(l2)
#> 7.29 kBLas listas pueden contener objetos complejos, por lo que no es posible elegir un único estilo visual que funcione para todas las listas. En general, dibujaré listas como vectores, usando colores para recordarle la jerarquía.
Las listas a veces se denominan vectores recursivos porque una lista puede contener otras listas. Esto los hace fundamentalmente diferentes de los vectores atómicos.
l3 <- list(list(list(1)))
str(l3)
#> List of 1
#> $ :List of 1
#> ..$ :List of 1
#> .. ..$ : num 1
c() combinará varias listas en una sola. Si se le da una combinación de vectores atómicos y listas, c() convertirá los vectores en listas antes de combinarlos. Compara los resultados de list() y c():
l4 <- list(list(1, 2), c(3, 4))
l5 <- c(list(1, 2), c(3, 4))
str(l4)
#> List of 2
#> $ :List of 2
#> ..$ : num 1
#> ..$ : num 2
#> $ : num [1:2] 3 4
str(l5)
#> List of 4
#> $ : num 1
#> $ : num 2
#> $ : num 3
#> $ : num 4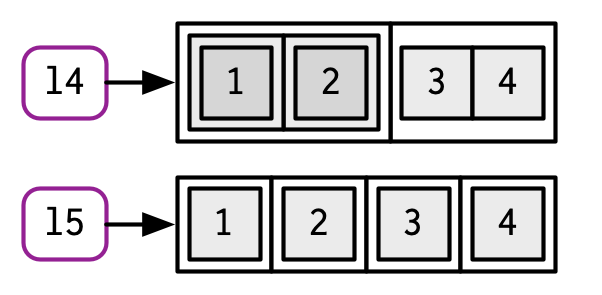
3.5.2 Pruebas y coerción
El typeof() una lista es list. Puede probar una lista con is.list() y obligar a una lista con as.list().
list(1:3)
#> [[1]]
#> [1] 1 2 3
as.list(1:3)
#> [[1]]
#> [1] 1
#>
#> [[2]]
#> [1] 2
#>
#> [[3]]
#> [1] 3Puedes convertir una lista en un vector atómico con unlist(). Las reglas para el tipo resultante son complejas, no están bien documentadas y no siempre son equivalentes a lo que obtendría con c().
3.5.3 Matrices y arreglos
Con vectores atómicos, el atributo de dimensión se usa comúnmente para crear matrices. Con las listas, el atributo de dimensión se puede usar para crear matrices de listas o arreglos de listas:
l <- list(1:3, "a", TRUE, 1.0)
dim(l) <- c(2, 2)
l
#> [,1] [,2]
#> [1,] integer,3 TRUE
#> [2,] "a" 1
l[[1, 1]]
#> [1] 1 2 3Estas estructuras de datos son relativamente esotéricas, pero pueden ser útiles si desea organizar objetos en una estructura similar a una cuadrícula. Por ejemplo, si está ejecutando modelos en una cuadrícula espacio-temporal, podría ser más intuitivo almacenar los modelos en una matriz 3D que coincida con la estructura de la cuadrícula.
3.5.4 Ejercicios
Enumere todas las formas en que una lista difiere de un vector atómico.
¿Por qué necesita usar
unlist()para convertir una lista en un vector atómico? ¿Por qué no funcionaas.vector()?Compare y contraste
c()yunlist()al combinar una fecha y una fecha y hora en un solo vector.
3.6 Data frames y tibbles
Los dos vectores S3 más importantes construidos sobre las listas son los data frames y los tibbles.
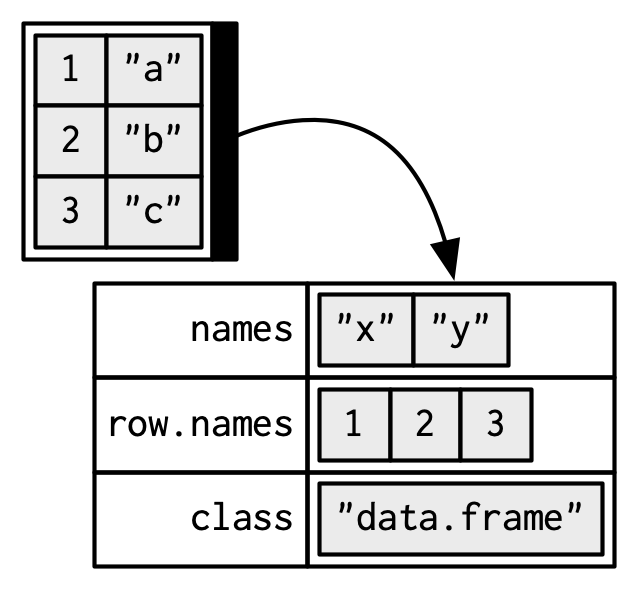
Si realiza análisis de datos en R, utilizará data frames. Un data frame es una lista con nombre de vectores con atributos para (columna) nombres, fila.nombres 12, y su clase, “data.frame”:
df1 <- data.frame(x = 1:3, y = letters[1:3])
typeof(df1)
#> [1] "list"
attributes(df1)
#> $names
#> [1] "x" "y"
#>
#> $class
#> [1] "data.frame"
#>
#> $row.names
#> [1] 1 2 3A diferencia de una lista regular, un data frame tiene una restricción adicional: la longitud de cada uno de sus vectores debe ser la misma. Esto le da a los data frames su estructura rectangular y explica por qué comparten las propiedades de las matrices y las listas:
Un data frame tiene
rownames()13 ycolnames(). Losnames()de un data frame son los nombres de las columnas.Un data frame tiene filas
nrow()y columnasncol(). Lalongitud()de un data frame da el número de columnas.
Los data frames son una de las ideas más grandes e importantes de R, y una de las cosas que diferencian a R de otros lenguajes de programación. Sin embargo, en los más de 20 años desde su creación, las formas en que las personas usan R han cambiado y algunas de las decisiones de diseño que tenían sentido en el momento en que se crearon los data frames ahora causan frustración.
Esta frustración condujo a la creación del tibble (Müller y Wickham 2018), una reinvención moderna del data frame. Los Tibbles están diseñados para ser (en la medida de lo posible) reemplazos directos de los data frames que solucionan esas frustraciones. Una forma concisa y divertida de resumir las principales diferencias es que los tibbles son vagos y hoscos: hacen menos y se quejan más. Verá lo que eso significa a medida que avance en esta sección.
Los tibbles son proporcionados por el paquete tibble y comparten la misma estructura que los data frames. La única diferencia es que el vector de clase es más largo e incluye tbl_df. Esto permite que los tibbles se comporten de manera diferente en las formas clave que discutiremos a continuación.
library(tibble)
df2 <- tibble(x = 1:3, y = letters[1:3])
typeof(df2)
#> [1] "list"
attributes(df2)
#> $class
#> [1] "tbl_df" "tbl" "data.frame"
#>
#> $row.names
#> [1] 1 2 3
#>
#> $names
#> [1] "x" "y"3.6.1 Creando
Creas un data frame proporcionando pares de vector de nombre a data.frame():
df <- data.frame(
x = 1:3,
y = c("a", "b", "c")
)
str(df)
#> 'data.frame': 3 obs. of 2 variables:
#> $ x: int 1 2 3
#> $ y: chr "a" "b" "c"Si está utilizando una versión de R anterior a la 4.0.0, tenga cuidado con la conversión predeterminada de cadenas a factores. Use stringsAsFactors = FALSE para suprimir esto y mantener los vectores de caracteres como vectores de caracteres:
df1 <- data.frame(
x = 1:3,
y = c("a", "b", "c"),
stringsAsFactors = FALSE
)
str(df1)
#> 'data.frame': 3 obs. of 2 variables:
#> $ x: int 1 2 3
#> $ y: chr "a" "b" "c"Crear un tibble es similar a crear un data frame. La diferencia entre los dos es que los tibbles nunca coaccionan su entrada (esta es una característica que los hace perezosos):
df2 <- tibble(
x = 1:3,
y = c("a", "b", "c")
)
str(df2)
#> tibble [3 × 2] (S3: tbl_df/tbl/data.frame)
#> $ x: int [1:3] 1 2 3
#> $ y: chr [1:3] "a" "b" "c"Además, mientras que los data frames transforman automáticamente los nombres no sintácticos (a menos que check.names = FALSE), los tibbles no lo hacen (aunque sí imprimen nombres no sintácticos rodeados por `).
names(data.frame(`1` = 1))
#> [1] "X1"
names(tibble(`1` = 1))
#> [1] "1"Si bien cada elemento de un data frame (o tibble) debe tener la misma longitud, tanto data.frame() como tibble() reciclarán entradas más cortas. Sin embargo, mientras que los data frames reciclan automáticamente las columnas que son múltiplos enteros de la columna más larga, los tibbles solo reciclan vectores de longitud uno.
data.frame(x = 1:4, y = 1:2)
#> x y
#> 1 1 1
#> 2 2 2
#> 3 3 1
#> 4 4 2
data.frame(x = 1:4, y = 1:3)
#> Error in data.frame(x = 1:4, y = 1:3): arguments imply differing number of rows: 4, 3
tibble(x = 1:4, y = 1)
#> # A tibble: 4 × 2
#> x y
#> <int> <dbl>
#> 1 1 1
#> 2 2 1
#> 3 3 1
#> 4 4 1
tibble(x = 1:4, y = 1:2)
#> Error in `tibble()`:
#> ! Tibble columns must have compatible sizes.
#> • Size 4: Existing data.
#> • Size 2: Column `y`.
#> ℹ Only values of size one are recycled.Hay una última diferencia: tibble() te permite referirte a las variables creadas durante la construcción:
tibble(
x = 1:3,
y = x * 2
)
#> # A tibble: 3 × 2
#> x y
#> <int> <dbl>
#> 1 1 2
#> 2 2 4
#> 3 3 6(Las entradas se evalúan de izquierda a derecha).
Al dibujar data frames y tibbles, en lugar de centrarse en los detalles de implementación, es decir, los atributos:
Los dibujaré de la misma manera que una lista con nombre, pero los ordenaré para enfatizar su estructura en columnas.
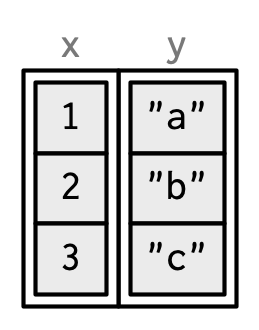
3.6.2 Nombres de filas
Los data frames le permiten etiquetar cada fila con un nombre, un vector de caracteres que contiene solo valores únicos:
df3 <- data.frame(
age = c(35, 27, 18),
hair = c("blond", "brown", "black"),
row.names = c("Bob", "Susan", "Sam")
)
df3
#> age hair
#> Bob 35 blond
#> Susan 27 brown
#> Sam 18 blackPuede obtener y establecer nombres de fila con rownames(), y puede usarlos para crear subconjuntos de filas:
rownames(df3)
#> [1] "Bob" "Susan" "Sam"
df3["Bob", ]
#> age hair
#> Bob 35 blondLos nombres de las filas surgen naturalmente si piensa en los data frames como estructuras 2D como matrices: las columnas (variables) tienen nombres, por lo que las filas (observaciones) también deberían tenerlos. La mayoría de las matrices son numéricas, por lo que es importante tener un lugar para almacenar etiquetas de caracteres. Pero esta analogía con las matrices es engañosa porque las matrices poseen una propiedad importante que los data frames no tienen: son transponibles. En las matrices, las filas y las columnas son intercambiables, y la transposición de una matriz da como resultado otra matriz (la transposición nuevamente da como resultado la matriz original). Sin embargo, con los data frames, las filas y las columnas no son intercambiables: la transposición de un data frame no es un data frame.
Hay tres razones por las que los nombres de las filas no son deseables:
Los metadatos son datos, por lo que almacenarlos de manera diferente al resto de los datos es fundamentalmente una mala idea. También significa que necesita aprender un nuevo conjunto de herramientas para trabajar con nombres de fila; no puede usar lo que ya sabe sobre la manipulación de columnas.
Los nombres de fila son una abstracción deficiente para etiquetar filas porque solo funcionan cuando una fila se puede identificar con una sola cadena. Esto falla en muchos casos, por ejemplo, cuando desea identificar una fila por un vector que no es un carácter (por ejemplo, un punto de tiempo), o con múltiples vectores (por ejemplo, posición, codificado por latitud y longitud).
Los nombres de las filas deben ser únicos, por lo que cualquier duplicación de filas (por ejemplo, de arranque) creará nuevos nombres de fila. Si desea hacer coincidir las filas de antes y después de la transformación, deberá realizar una complicada cirugía de hilo.
df3[c(1, 1, 1), ] #> age hair #> Bob 35 blond #> Bob.1 35 blond #> Bob.2 35 blond
Por estas razones, los tibbles no admiten nombres de fila. En cambio, el paquete tibble proporciona herramientas para convertir fácilmente los nombres de las filas en una columna regular con rownames_to_column(), o el argumento rownames en as_tibble():
as_tibble(df3, rownames = "name")
#> # A tibble: 3 × 3
#> name age hair
#> <chr> <dbl> <chr>
#> 1 Bob 35 blond
#> 2 Susan 27 brown
#> 3 Sam 18 black3.6.3 Imprimir
Una de las diferencias más obvias entre tibbles y data frames es cómo se imprimen. Supongo que ya está familiarizado con la forma en que se imprimen los data frames, por lo que aquí resaltaré algunas de las mayores diferencias utilizando un conjunto de datos de ejemplo incluido en el paquete dplyr:
dplyr::starwars
#> # A tibble: 87 × 14
#> name height mass hair_color skin_color eye_color birth_year sex gender
#> <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
#> 1 Luke Sk… 172 77 blond fair blue 19 male mascu…
#> 2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
#> 3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
#> 4 Darth V… 202 136 none white yellow 41.9 male mascu…
#> 5 Leia Or… 150 49 brown light brown 19 fema… femin…
#> 6 Owen La… 178 120 brown, gr… light blue 52 male mascu…
#> 7 Beru Wh… 165 75 brown light blue 47 fema… femin…
#> 8 R5-D4 97 32 <NA> white, red red NA none mascu…
#> 9 Biggs D… 183 84 black light brown 24 male mascu…
#> 10 Obi-Wan… 182 77 auburn, w… fair blue-gray 57 male mascu…
#> # ℹ 77 more rows
#> # ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
#> # vehicles <list>, starships <list>Tibbles solo muestra las primeras 10 filas y todas las columnas que caben en la pantalla. Las columnas adicionales se muestran en la parte inferior.
Cada columna está etiquetada con su tipo, abreviado en tres o cuatro letras.
Las columnas anchas se truncan para evitar que una sola cadena larga ocupe una fila completa. (Esto todavía es un trabajo en progreso: es un compromiso complicado entre mostrar tantas columnas como sea posible y mostrar las columnas en su totalidad).
Cuando se usa en entornos de consola que lo admiten, el color se usa juiciosamente para resaltar información importante y restar énfasis a los detalles complementarios.
3.6.4 Subconjunto
Como aprenderá en el Capítulo 4, puede crear un subconjunto de un data frame o un tibble como una estructura 1D (donde se comporta como una lista), o una estructura 2D (donde se comporta como una matriz).
En mi opinión, los data frames tienen dos comportamientos de subconjunto no deseados:
Cuando crea subconjuntos de columnas con
df[, vars], obtendrá un vector sivarsselecciona una variable; de lo contrario, obtendrá un data frame. Esta es una fuente frecuente de errores cuando se usa[en una función, a menos que siempre recuerde usardf[, vars, drop = FALSE].Cuando intenta extraer una sola columna con
df$xy no hay ninguna columnax, un data frame seleccionará cualquier variable que comience conx. Si ninguna variable comienza conx,df$xdevolveráNULL. Esto facilita seleccionar la variable incorrecta o seleccionar una variable que no existe14.
Tibbles modifica estos comportamientos para que un [ siempre devuelva un tibble, y un $ no haga coincidencias parciales y advierta si no puede encontrar una variable (esto es lo que hace que Tibbles sea hosco).
df1 <- data.frame(xyz = "a")
df2 <- tibble(xyz = "a")
str(df1$x)
#> chr "a"
str(df2$x)
#> Warning: Unknown or uninitialised column: `x`.
#> NULLLa insistencia de un tibble en devolver un data frame de [ puede causar problemas con el código heredado, que a menudo usa df[, "col"] para extraer una sola columna. Si desea una sola columna, le recomiendo usar df[["col"]]. Esto comunica claramente su intención y funciona tanto con data frames como con tibbles.
3.6.5 Pruebas y coacción
Para verificar si un objeto es un data frame o tibble, use is.data.frame():
is.data.frame(df1)
#> [1] TRUE
is.data.frame(df2)
#> [1] TRUEPor lo general, no debería importar si tiene un tibble o un data frame, pero si necesita estar seguro, use is_tibble():
is_tibble(df1)
#> [1] FALSE
is_tibble(df2)
#> [1] TRUEPuede convertir un objeto en un data frame con as.data.frame() o en un tibble con as_tibble().
3.6.6 Columnas de lista
Dado que un data frame es una lista de vectores, es posible que un data frame tenga una columna que sea una lista. Esto es muy útil porque una lista puede contener cualquier otro objeto: esto significa que puede colocar cualquier objeto en un data frame. Esto le permite mantener los objetos relacionados juntos en una fila, sin importar cuán complejos sean los objetos individuales. Puede ver una aplicación de esto en el capítulo “Muchos modelos” de R para la Ciencia de Datos, http://r4ds.had.co.nz/many-models.html.
Las columnas de lista están permitidas en data frames, pero debe hacer un poco de trabajo adicional agregando la columna de lista después de la creación o envolviendo la lista en I()15.
df <- data.frame(x = 1:3)
df$y <- list(1:2, 1:3, 1:4)
data.frame(
x = 1:3,
y = I(list(1:2, 1:3, 1:4))
)
#> x y
#> 1 1 1, 2
#> 2 2 1, 2, 3
#> 3 3 1, 2, 3, 4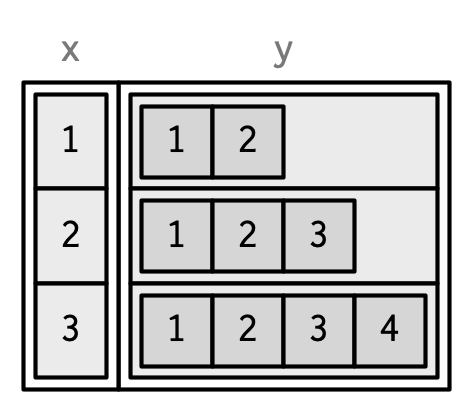
Las columnas de lista son más fáciles de usar con tibbles porque se pueden incluir directamente dentro de tibble() y se imprimirán ordenadamente:
tibble(
x = 1:3,
y = list(1:2, 1:3, 1:4)
)
#> # A tibble: 3 × 2
#> x y
#> <int> <list>
#> 1 1 <int [2]>
#> 2 2 <int [3]>
#> 3 3 <int [4]>3.6.7 Columnas de matriz y data frame
Siempre que el número de filas coincida con el data frame, también es posible tener una matriz o arreglo como columna de un data frame. (Esto requiere una ligera extensión a nuestra definición de data frame: no es la longitud, length(), de cada columna lo que debe ser igual, sino el NROW().) En cuanto a las columnas de lista, debe agregarlas después de la creación o envolverlas en I().
dfm <- data.frame(
x = 1:3 * 10
)
dfm$y <- matrix(1:9, nrow = 3)
dfm$z <- data.frame(a = 3:1, b = letters[1:3], stringsAsFactors = FALSE)
str(dfm)
#> 'data.frame': 3 obs. of 3 variables:
#> $ x: num 10 20 30
#> $ y: int [1:3, 1:3] 1 2 3 4 5 6 7 8 9
#> $ z:'data.frame': 3 obs. of 2 variables:
#> ..$ a: int 3 2 1
#> ..$ b: chr "a" "b" "c"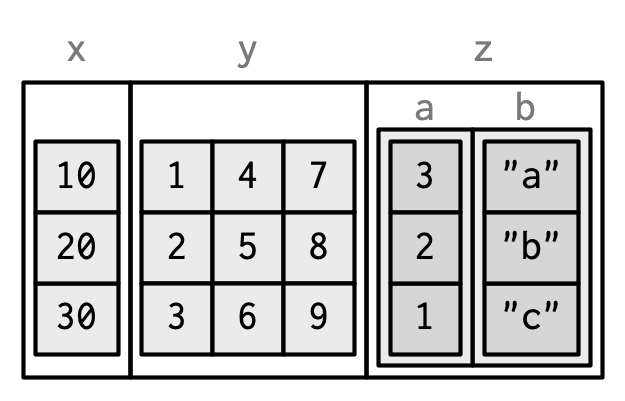
Las columnas de matrices y data frames requieren un poco de precaución. Muchas funciones que trabajan con data frames asumen que todas las columnas son vectores. Además, la pantalla impresa puede resultar confusa.
dfm[1, ]
#> x y.1 y.2 y.3 z.a z.b
#> 1 10 1 4 7 3 a3.6.8 Ejercicios
¿Puede tener un data frame con cero filas? ¿Qué pasa con las columnas cero?
¿Qué sucede si intenta establecer nombres de fila que no son únicos?
Si
dfes un data frame, ¿qué puede decir acerca det(df)yt(t(df))? Realice algunos experimentos, asegurándose de probar diferentes tipos de columnas.¿Qué hace
as.matrix()cuando se aplica a un data frame con columnas de diferentes tipos? ¿En qué se diferencia dedata.matrix()?
3.7 NULL
Para terminar este capítulo, quiero hablar sobre una última estructura de datos importante que está estrechamente relacionada con los vectores: NULL. NULL es especial porque tiene un tipo único, siempre tiene una longitud cero y no puede tener ningún atributo:
typeof(NULL)
#> [1] "NULL"
length(NULL)
#> [1] 0
x <- NULL
attr(x, "y") <- 1
#> Error in attr(x, "y") <- 1: attempt to set an attribute on NULLPuedes probar NULLs con is.null():
is.null(NULL)
#> [1] TRUEHay dos usos comunes de NULL:
Representar un vector vacío (un vector de longitud cero) de tipo arbitrario. Por ejemplo, si usas
c()pero no incluyes ningún argumento, obtienesNULL, y concatenarNULLa un vector lo dejará sin cambios:c() #> NULLPara representar un vector ausente. Por ejemplo,
NULLa menudo se usa como argumento de función predeterminado, cuando el argumento es opcional pero el valor predeterminado requiere algún cálculo (consulte la Sección 6.5.3 para obtener más información al respecto). Contraste esto conNAque se usa para indicar que un elemento de un vector está ausente.
Si está familiarizado con SQL, conocerá el NULL relacional y puede esperar que sea igual que el de R. Sin embargo, la base de datos NULL es en realidad equivalente a NA de R.
3.8 Respuestas de la prueba
Los cuatro tipos comunes de vectores atómicos son lógicos, enteros, dobles y de carácter. Los dos tipos más raros son complejos y crudos.
Los atributos le permiten asociar metadatos adicionales arbitrarios a cualquier objeto. Puede obtener y establecer atributos individuales con
attr(x, "y")yattr(x, "y") <- value; o puede obtener y establecer todos los atributos a la vez conattributes().Los elementos de una lista pueden ser de cualquier tipo (incluso una lista); los elementos de un vector atómico son todos del mismo tipo. De manera similar, todos los elementos de una matriz deben ser del mismo tipo; en un data frame, diferentes columnas pueden tener diferentes tipos.
Puede crear una matriz de lista asignando dimensiones a una lista. Puede convertir una matriz en una columna de un data frame con
df$x <- matrix(), o usandoI()al crear un nuevo data framedata.frame(x = I(matrix())).Los Tibbles tienen un método de impresión mejorado, nunca obligan a las cadenas a factores y proporcionan métodos de subconjunto más estrictos.
En conjunto, todos los demás tipos de datos se conocen como tipos de “nodo”, que incluyen cosas como funciones y entornos. Lo más probable es que te encuentres con este término altamente técnico cuando uses
gc(): la “N” enNcellssignifica nodos y la “V” enVcellssignifica vectores.↩︎Algunos lugares en la documentación de R llaman a listas de vectores genéricos para enfatizar su diferencia con los vectores atómicos.↩︎
Esta es una ligera simplificación ya que R no usa “numérico” de manera consistente, a lo que volveremos en la Sección 12.3.1.↩︎
Técnicamente, el lenguaje R no posee escalares. Todo lo que parece un escalar es en realidad un vector de longitud uno. Esta es principalmente una distinción teórica, pero significa que expresiones como
1[1]funcionan.↩︎Lno es intuitivo, y puede que te preguntes de dónde viene. En el momento en que se agregóLa R, el tipo de entero de R era equivalente a un entero largo en C, y el código C podía usar un sufijo deloLpara obligar a un número a ser un entero largo. Se decidió quelera demasiado similar visualmente ai(usado para números complejos en R), dejandoL.↩︎Es posible que haya oído hablar de las funciones
mode()ystorage.mode()relacionadas. No los uses: existen solo por compatibilidad con S.↩︎Bueno, técnicamente, cinco si incluimos
NA_complex_, pero ignoramos este tipo “raro”, como se mencionó anteriormente.↩︎Los atributos se comportan como listas con nombre, pero en realidad son listas de pares. Las listas de pares son funcionalmente indistinguibles de las listas, pero son profundamente diferentes bajo el capó. Aprenderá más sobre ellos en la Sección 18.6.1.↩︎
El tidyverse nunca fuerza automáticamente a los personajes a factores, y proporciona el paquete forcats (Wickham 2018) específicamente para trabajar con factores.↩︎
Esta fecha especial se conoce como la Época Unix.↩︎
tidyverse proporciona el paquete lubridate (Grolemund y Wickham 2011) para trabajar con fechas y horas. Proporciona una serie de útiles ayudantes que funcionan con el tipo POSIXct base.↩︎
Los nombres de fila son una de las estructuras de datos más sorprendentemente complejas en R. También han sido una fuente persistente de problemas de rendimiento a lo largo de los años. La implementación más sencilla es un vector de caracteres o entero, con un elemento para cada fila. Pero también hay una representación compacta para nombres de fila “automáticos” (enteros consecutivos), creada por
.set_row_names(). R 3.5 tiene una forma especial de diferir la conversión de enteros a caracteres que está diseñada específicamente para acelerarlm(); consulte https://svn.r-project.org/R/branches/ALTREP/ALTREP.html#deferred_string_conversions para obtener detalles.↩︎técnicamente, se recomienda usar
row.names(), norownames()con data frames, pero esta distinción rara vez es importante.↩︎Podemos hacer que R advierta sobre este comportamiento (llamado coincidencia parcial) configurando
options(warnPartialMatchDollar = TRUE).↩︎I()es la abreviatura de identidad y se usa a menudo para indicar que una entrada debe dejarse como está y no transformarse automáticamente.↩︎