5 Ordenando datos
5.1 Introducción
“Las familias felices son todas iguales; cada familia infeliz es infeliz a su manera.”
— Leo Tolstoy
“Los conjuntos de datos ordenados son todos iguales, pero cada conjunto de datos desordenado es desordenado a su manera.”
— Hadley Wickham
En este capítulo, aprenderá una forma consistente de organizar sus datos en R utilizando un sistema llamado tidy data (datos ordenados). Obtener sus datos en este formato requiere algo de trabajo por adelantado, pero ese trabajo vale la pena a largo plazo. Una vez que tenga datos ordenados y las herramientas ordenadas proporcionadas por los paquetes en tidyverse, pasará mucho menos tiempo pasando datos de una representación a otra, lo que le permitirá dedicar más tiempo a las preguntas de datos que le interesan.
En este capítulo, primero aprenderá la definición de datos ordenados y la verá aplicada a un conjunto de datos simple. Luego nos sumergiremos en la herramienta principal que usará para ordenar los datos: pivotar. Pivotar le permite cambiar la forma de sus datos sin cambiar ninguno de los valores.
5.1.1 Requisitos previos
En este capítulo, nos centraremos en tidyr, un paquete que proporciona un montón de herramientas para ayudar a ordenar sus desordenados conjuntos de datos. tidyr es miembro del núcleo tidyverse.
A partir de este capítulo, suprimiremos el mensaje de carga de library(tidyverse).
5.2 Tidy data
Puede representar los mismos datos subyacentes de varias formas. El siguiente ejemplo muestra los mismos datos organizados de tres maneras diferentes. Cada conjunto de datos muestra los mismos valores de cuatro variables: país (country), año (year), población (population) y número de casos (cases) documentados de TB (tuberculosis), pero cada conjunto de datos organiza los valores de manera diferente.
table1
#> # A tibble: 6 × 4
#> country year cases population
#> <chr> <dbl> <dbl> <dbl>
#> 1 Afghanistan 1999 745 19987071
#> 2 Afghanistan 2000 2666 20595360
#> 3 Brazil 1999 37737 172006362
#> 4 Brazil 2000 80488 174504898
#> 5 China 1999 212258 1272915272
#> 6 China 2000 213766 1280428583
table2
#> # A tibble: 12 × 4
#> country year type count
#> <chr> <dbl> <chr> <dbl>
#> 1 Afghanistan 1999 cases 745
#> 2 Afghanistan 1999 population 19987071
#> 3 Afghanistan 2000 cases 2666
#> 4 Afghanistan 2000 population 20595360
#> 5 Brazil 1999 cases 37737
#> 6 Brazil 1999 population 172006362
#> # ℹ 6 more rows
table3
#> # A tibble: 6 × 3
#> country year rate
#> <chr> <dbl> <chr>
#> 1 Afghanistan 1999 745/19987071
#> 2 Afghanistan 2000 2666/20595360
#> 3 Brazil 1999 37737/172006362
#> 4 Brazil 2000 80488/174504898
#> 5 China 1999 212258/1272915272
#> 6 China 2000 213766/1280428583Todas estas son representaciones de los mismos datos subyacentes, pero no son igualmente fáciles de usar. Uno de ellos, table1, será mucho más fácil de trabajar dentro del tidyverse porque esta ordenada.
Hay tres reglas interrelacionadas que hacen que un conjunto de datos esté ordenado:
- Cada variable es una columna; cada columna es una variable.
- Cada observación es una fila; cada fila es una observación.
- Cada valor es una celda; cada celda es un valor único.
Figura 5.1 muestra las reglas visualmente.

¿Por qué asegurarse de que sus datos estén ordenados? Hay dos ventajas principales:
Hay una ventaja general en elegir una forma consistente de almacenar datos. Si tiene una estructura de datos consistente, es más fácil aprender las herramientas que funcionan con ella porque tienen una uniformidad subyacente.
Hay una ventaja específica en colocar variables en columnas porque permite que brille la naturaleza vectorizada de R. Como aprendió en Sección 3.3.1 y Sección 3.5.2, la mayoría de las funciones integradas de R funcionan con vectores de valores. Eso hace que la transformación de datos ordenados se sienta particularmente natural.
dplyr, ggplot2 y todos los demás paquetes en tidyverse están diseñados para funcionar con datos ordenados. Aquí hay algunos pequeños ejemplos que muestran cómo podría trabajar con table1.
# Tasa de cálculo por 10.000
table1 |>
mutate(rate = cases / population * 10000)
#> # A tibble: 6 × 5
#> country year cases population rate
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 Afghanistan 1999 745 19987071 0.373
#> 2 Afghanistan 2000 2666 20595360 1.29
#> 3 Brazil 1999 37737 172006362 2.19
#> 4 Brazil 2000 80488 174504898 4.61
#> 5 China 1999 212258 1272915272 1.67
#> 6 China 2000 213766 1280428583 1.67
# Calcular casos por año
table1 |>
group_by(year) |>
summarize(total_cases = sum(cases))
#> # A tibble: 2 × 2
#> year total_cases
#> <dbl> <dbl>
#> 1 1999 250740
#> 2 2000 296920
# Visualice los cambios a lo largo del tiempo
ggplot(table1, aes(x = year, y = cases)) +
geom_line(aes(group = country), color = "grey50") +
geom_point(aes(color = country, shape = country)) +
scale_x_continuous(breaks = c(1999, 2000)) # x-axis breaks at 1999 and 2000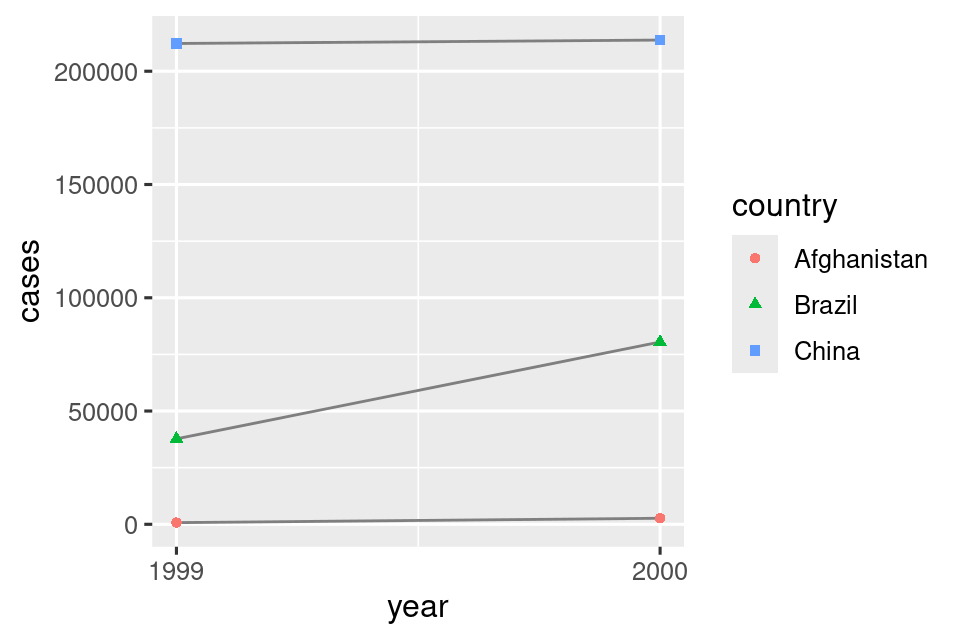
5.2.1 Ejercicios
Para cada una de las tablas de muestra, describa lo que representa cada observación y cada columna.
-
Haz un bosquejo del proceso que usarías para calcular la
ratedesdetable2. Deberá realizar cuatro operaciones:- Extraiga el número de casos de TB por país por año.
- Extraiga la población coincidente por país por año.
- Divida los casos por la población y multiplique por 10000.
- Guárdelo nuevamente en el lugar apropiado.
Todavía no ha aprendido todas las funciones que necesitaría para realizar estas operaciones, pero aún debería poder pensar en las transformaciones que necesitaría.
5.3 Alargar datos
Los principios de los datos ordenados pueden parecer tan obvios que se pregunta si alguna vez se encontrará con un conjunto de datos que no esté ordenado. Desafortunadamente, sin embargo, la mayoría de los datos reales están desordenados. Hay dos razones principales:
Los datos a menudo se organizan para facilitar algún objetivo que no sea el análisis. Por ejemplo, es común que los datos estén estructurados para facilitar la entrada de datos, no el análisis.
La mayoría de las personas no están familiarizadas con los principios de los datos ordenados, y es difícil derivarlos usted mismo a menos que pase mucho tiempo trabajando con datos.
Esto significa que la mayoría de los análisis reales requerirán al menos un poco de limpieza. Comenzará averiguando cuáles son las variables y observaciones subyacentes. A veces esto es fácil; otras veces necesitará consultar con las personas que generaron originalmente los datos. A continuación, pivotará sus datos en una forma ordenada, con variables en las columnas y observaciones en las filas.
tidyr proporciona dos funciones para pivotar datos: pivot_longer() y pivot_wider(). Empezaremos con pivot_longer() porque es el caso más común. Veamos unos ejemplos.
5.3.1 Datos en nombres de columna
El conjunto de datos billboard registra el rango de las canciones en la cartelera en el año 2000:
billboard
#> # A tibble: 317 × 79
#> artist track date.entered wk1 wk2 wk3 wk4 wk5
#> <chr> <chr> <date> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 2 Pac Baby Don't Cry (Ke… 2000-02-26 87 82 72 77 87
#> 2 2Ge+her The Hardest Part O… 2000-09-02 91 87 92 NA NA
#> 3 3 Doors Down Kryptonite 2000-04-08 81 70 68 67 66
#> 4 3 Doors Down Loser 2000-10-21 76 76 72 69 67
#> 5 504 Boyz Wobble Wobble 2000-04-15 57 34 25 17 17
#> 6 98^0 Give Me Just One N… 2000-08-19 51 39 34 26 26
#> # ℹ 311 more rows
#> # ℹ 71 more variables: wk6 <dbl>, wk7 <dbl>, wk8 <dbl>, wk9 <dbl>, …En este conjunto de datos, cada observación es una canción. Las primeras tres columnas (artist, track y date.entered) son variables que describen la canción. Luego tenemos 76 columnas (wk1-wk76) que describen el rango de la canción en cada semana1. Aquí, los nombres de las columnas son una variable (la semana) y los valores de las celdas son otra (el rango).
Para ordenar estos datos, usaremos pivot_longer():
billboard |>
pivot_longer(
cols = starts_with("wk"),
names_to = "week",
values_to = "rank"
)
#> # A tibble: 24,092 × 5
#> artist track date.entered week rank
#> <chr> <chr> <date> <chr> <dbl>
#> 1 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk1 87
#> 2 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk2 82
#> 3 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk3 72
#> 4 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk4 77
#> 5 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk5 87
#> 6 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk6 94
#> 7 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk7 99
#> 8 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk8 NA
#> 9 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk9 NA
#> 10 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk10 NA
#> # ℹ 24,082 more rowsDespués de los datos, hay tres argumentos clave:
-
colsespecifica qué columnas deben girarse, es decir, qué columnas no son variables. Este argumento usa la misma sintaxis queselect(), así que aquí podríamos usar!c(artist, track, date.entered)ostarts_with("wk"). -
names_tonombra la variable almacenada en los nombres de columna, llamamos a esa variableweek. -
values_tonombra la variable almacenada en los valores de celda, llamamos a esa variablerank.
Tenga en cuenta que en el código se citan "week" y "rank" porque son variables nuevas que estamos creando, aún no existen en los datos cuando ejecutamos la llamada pivot_longer().
Ahora dirijamos nuestra atención al marco de datos más largo resultante. ¿Qué sucede si una canción está en el top 100 durante menos de 76 semanas? Tome “Baby Don’t Cry” de 2 Pac, por ejemplo. El resultado anterior sugiere que estuvo solo en el top 100 durante 7 semanas, y todas las semanas restantes se completan con valores faltantes. Estas NA no representan realmente observaciones desconocidas; están obligados a existir por la estructura del conjunto de datos 2, por lo que podemos pedirle a pivot_longer() que se deshaga de ellos configurando values_drop_na = TRUE:
billboard |>
pivot_longer(
cols = starts_with("wk"),
names_to = "week",
values_to = "rank",
values_drop_na = TRUE
)
#> # A tibble: 5,307 × 5
#> artist track date.entered week rank
#> <chr> <chr> <date> <chr> <dbl>
#> 1 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk1 87
#> 2 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk2 82
#> 3 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk3 72
#> 4 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk4 77
#> 5 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk5 87
#> 6 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk6 94
#> # ℹ 5,301 more rowsEl número de filas ahora es mucho menor, lo que indica que se eliminaron muchas filas con NA.
También puede preguntarse qué sucede si una canción está en el top 100 durante más de 76 semanas. No podemos decirlo a partir de estos datos, pero puede suponer que se agregarán columnas adicionales wk77, wk78, … al conjunto de datos.
Estos datos ahora están ordenados, pero podríamos hacer que el cálculo futuro sea un poco más fácil al convertir week en un número usando mutate() y readr::parse_number(). parse_number() es una función útil que extraerá el primer número de una cadena, ignorando el resto del texto.
billboard_longer <- billboard |>
pivot_longer(
cols = starts_with("wk"),
names_to = "week",
values_to = "rank",
values_drop_na = TRUE
) |>
mutate(
week = parse_number(week)
)
billboard_longer
#> # A tibble: 5,307 × 5
#> artist track date.entered week rank
#> <chr> <chr> <date> <dbl> <dbl>
#> 1 2 Pac Baby Don't Cry (Keep... 2000-02-26 1 87
#> 2 2 Pac Baby Don't Cry (Keep... 2000-02-26 2 82
#> 3 2 Pac Baby Don't Cry (Keep... 2000-02-26 3 72
#> 4 2 Pac Baby Don't Cry (Keep... 2000-02-26 4 77
#> 5 2 Pac Baby Don't Cry (Keep... 2000-02-26 5 87
#> 6 2 Pac Baby Don't Cry (Keep... 2000-02-26 6 94
#> # ℹ 5,301 more rowsAhora que tenemos todos los números de semana en una variable y todos los valores de clasificación en otra, estamos en una buena posición para visualizar cómo varían las clasificaciones de las canciones con el tiempo. El código se muestra a continuación y el resultado está en Figura 5.2. Podemos ver que muy pocas canciones permanecen en el top 100 por más de 20 semanas.
billboard_longer |>
ggplot(aes(x = week, y = rank, group = track)) +
geom_line(alpha = 0.25) +
scale_y_reverse()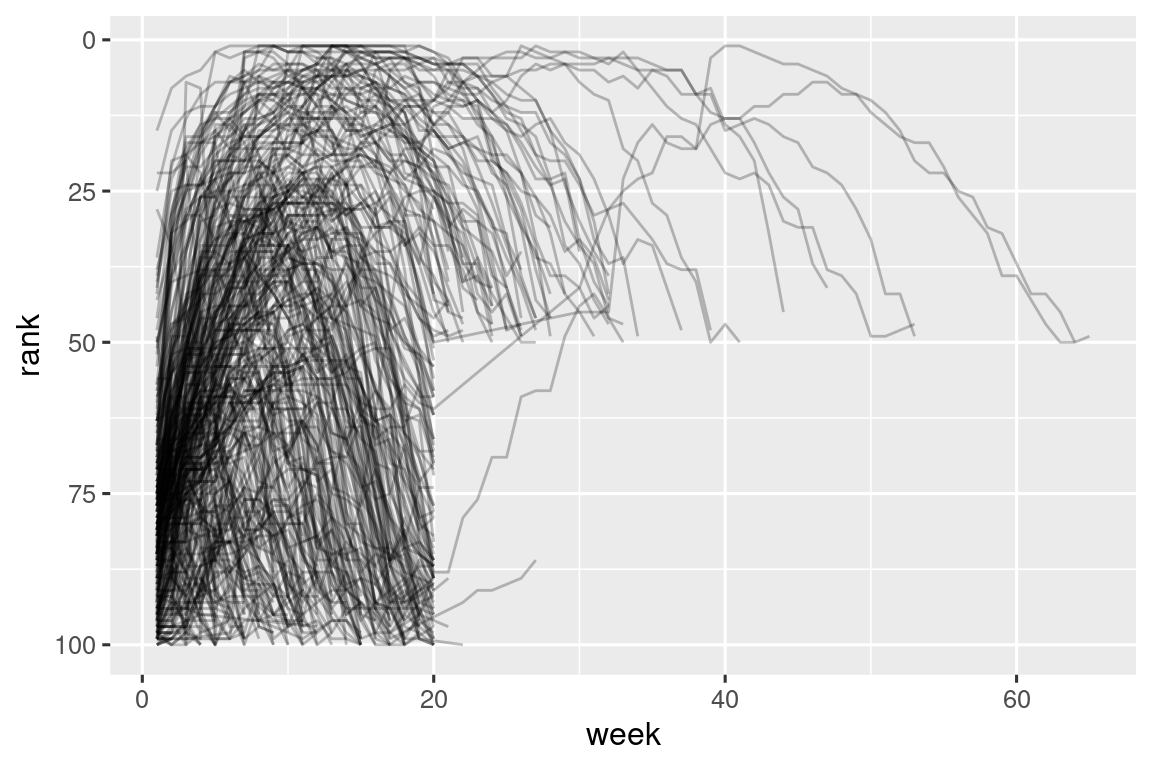
5.3.2 ¿Cómo funciona el pivoteo?
Ahora que ha visto cómo podemos usar el pivote para remodelar nuestros datos, tomemos un poco de tiempo para ganar algo de intuición sobre lo que hace el pivote con los datos. Comencemos con un conjunto de datos muy simple para que sea más fácil ver lo que está sucediendo. Supongamos que tenemos tres pacientes con ids A, B y C, y tomamos dos medidas de presión arterial en cada paciente. Crearemos los datos con tribble(), una función útil para construir pequeños tibbles a mano:
df <- tribble(
~id, ~bp1, ~bp2,
"A", 100, 120,
"B", 140, 115,
"C", 120, 125
)Queremos que nuestro nuevo conjunto de datos tenga tres variables: id (ya existe), measurement (los nombres de las columnas) y value (los valores de las celdas). Para lograr esto, necesitamos pivotear df por más tiempo:
df |>
pivot_longer(
cols = bp1:bp2,
names_to = "measurement",
values_to = "value"
)
#> # A tibble: 6 × 3
#> id measurement value
#> <chr> <chr> <dbl>
#> 1 A bp1 100
#> 2 A bp2 120
#> 3 B bp1 140
#> 4 B bp2 115
#> 5 C bp1 120
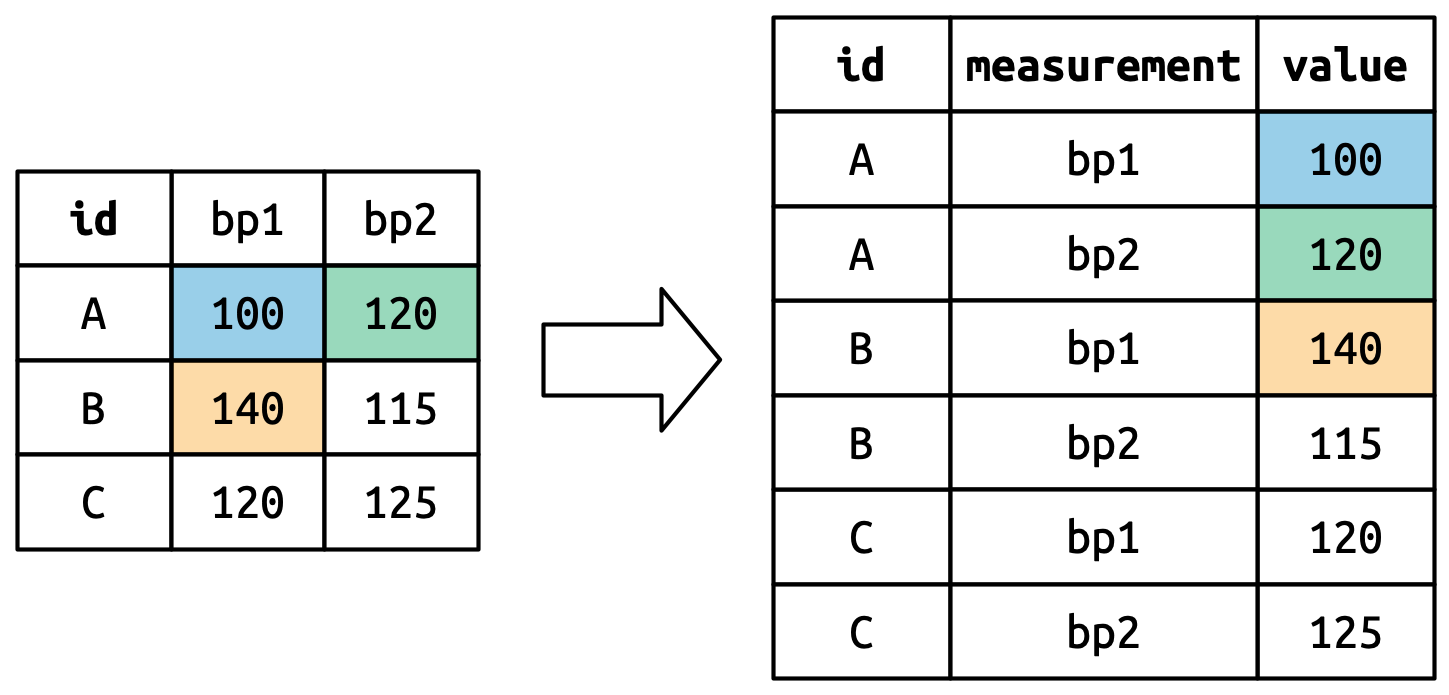
#> 6 C bp2 125¿Cómo funciona la remodelación? Es más fácil verlo si lo pensamos columna por columna. Como se muestra en Figura 5.3, los valores en la columna que ya era una variable en el conjunto de datos original (id) deben repetirse, una vez por cada columna que se pivote.
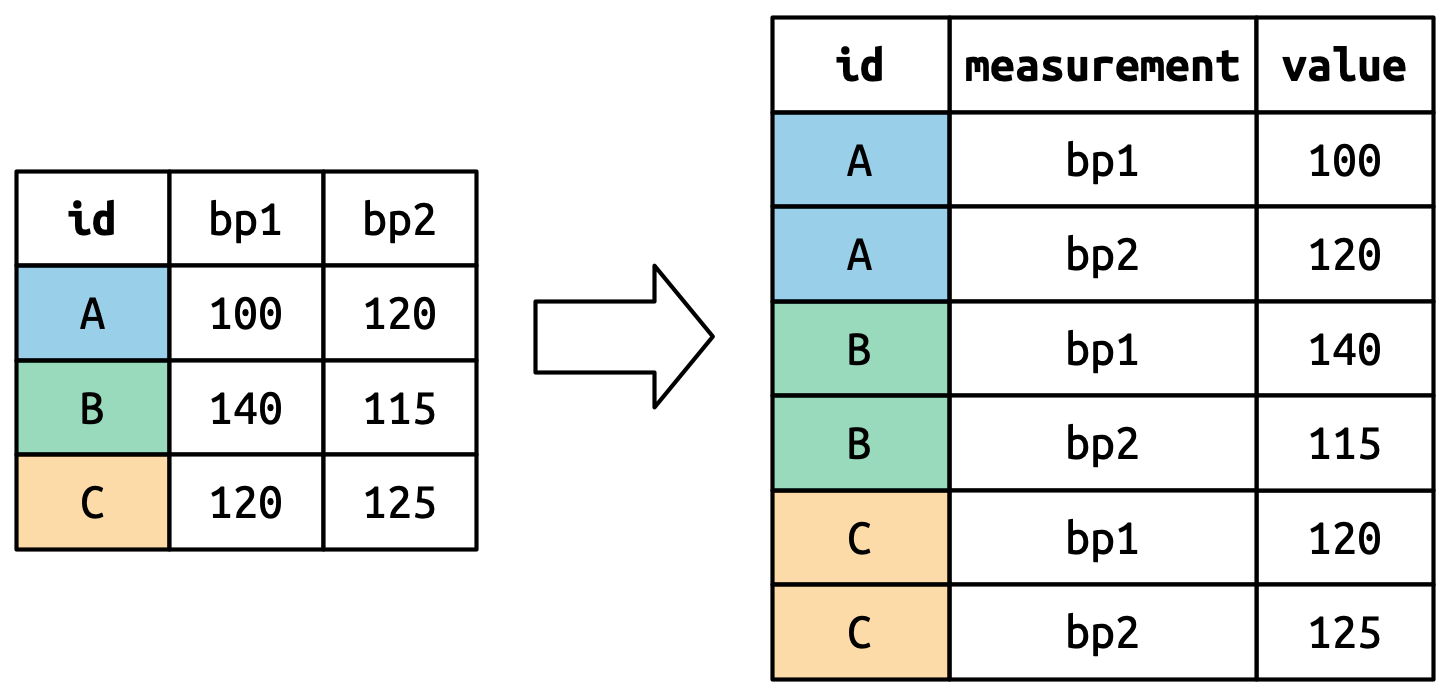
Los nombres de las columnas se convierten en valores en una nueva variable, cuyo nombre es definido por names_to, como se muestra en Figura 5.4. Deben repetirse una vez para cada fila en el conjunto de datos original.
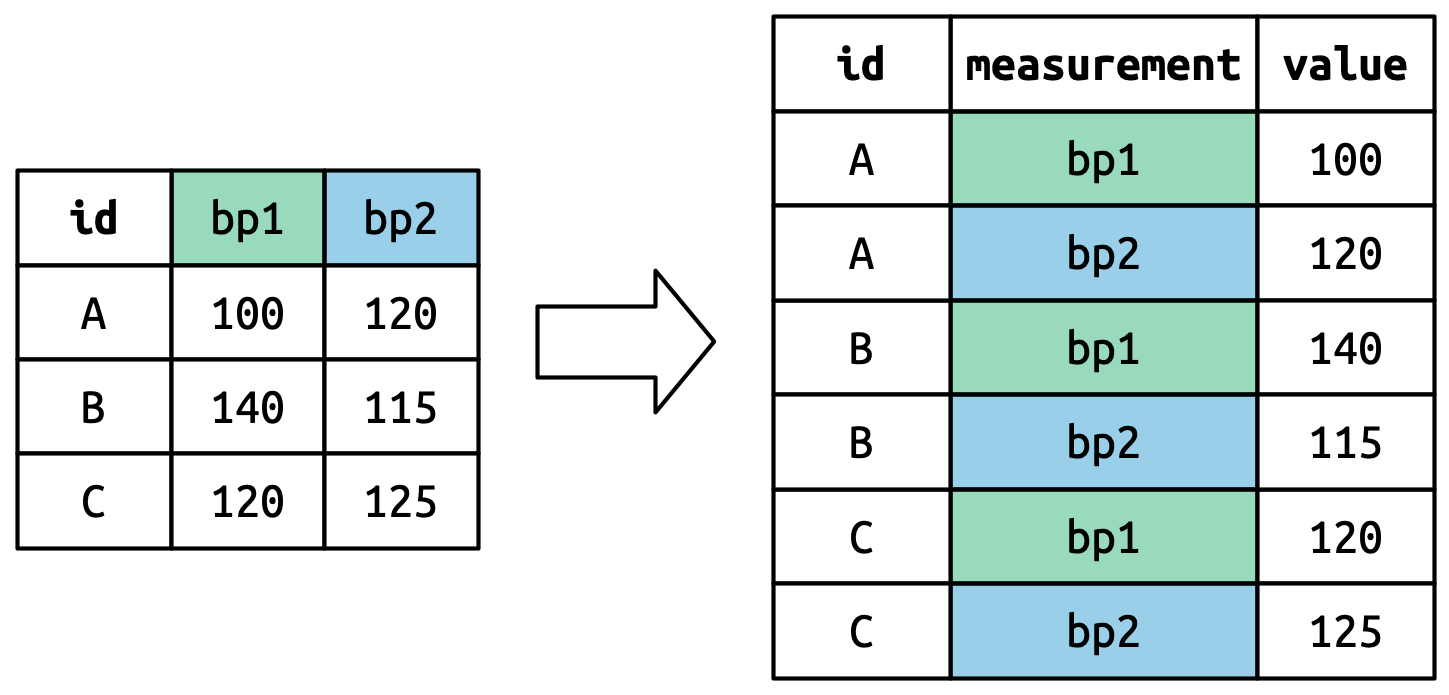
Los valores de celda también se convierten en valores en una nueva variable, con un nombre definido por values_to. Se desenrollan fila por fila. Figura 5.5 ilustra el proceso.
5.3.3 Muchas variables en los nombres de las columnas
Una situación más desafiante ocurre cuando tiene múltiples piezas de información abarrotadas en los nombres de las columnas y desea almacenarlas en nuevas variables separadas. Por ejemplo, tome el conjunto de datos who2, la fuente de table_1 y amigos que vió con anterioridad:
who2
#> # A tibble: 7,240 × 58
#> country year sp_m_014 sp_m_1524 sp_m_2534 sp_m_3544 sp_m_4554
#> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Afghanistan 1980 NA NA NA NA NA
#> 2 Afghanistan 1981 NA NA NA NA NA
#> 3 Afghanistan 1982 NA NA NA NA NA
#> 4 Afghanistan 1983 NA NA NA NA NA
#> 5 Afghanistan 1984 NA NA NA NA NA
#> 6 Afghanistan 1985 NA NA NA NA NA
#> # ℹ 7,234 more rows
#> # ℹ 51 more variables: sp_m_5564 <dbl>, sp_m_65 <dbl>, sp_f_014 <dbl>, …Este conjunto de datos, recopilado por la Organización Mundial de la Salud, registra información sobre diagnósticos de tuberculosis. Hay dos columnas que ya son variables y son fáciles de interpretar: country y year. Les siguen 56 columnas como sp_m_014, ep_m_4554 y rel_m_3544. Si observa estas columnas durante el tiempo suficiente, notará que hay un patrón. Cada nombre de columna se compone de tres piezas separadas por _. La primera pieza, sp/rel/ep, describe el método utilizado para el diagnóstico, la segunda pieza, m/f es el género (codificado como una variable binaria en este conjunto de datos) , y la tercera pieza, 014/1524/2534/3544/4554/5564/``65 es el rango de edad (014 representa 0-14, por ejemplo).
Entonces, en este caso, tenemos seis piezas de información registradas en who2: el país y el año (ya columnas); el método de diagnóstico, la categoría de género y la categoría de rango de edad (contenidas en los otros nombres de columna); y el recuento de pacientes en esa categoría (valores de celda). Para organizar estas seis piezas de información en seis columnas separadas, usamos pivot_longer() con un vector de nombres de columna para names_to e instructores para dividir los nombres de las variables originales en partes para names_sep, así como un nombre de columna para values_to:
who2 |>
pivot_longer(
cols = !(country:year),
names_to = c("diagnosis", "gender", "age"),
names_sep = "_",
values_to = "count"
)
#> # A tibble: 405,440 × 6
#> country year diagnosis gender age count
#> <chr> <dbl> <chr> <chr> <chr> <dbl>
#> 1 Afghanistan 1980 sp m 014 NA
#> 2 Afghanistan 1980 sp m 1524 NA
#> 3 Afghanistan 1980 sp m 2534 NA
#> 4 Afghanistan 1980 sp m 3544 NA
#> 5 Afghanistan 1980 sp m 4554 NA
#> 6 Afghanistan 1980 sp m 5564 NA
#> # ℹ 405,434 more rowsUna alternativa a names_sep es names_pattern, que puede usar para extraer variables de escenarios de nombres más complicados, una vez que haya aprendido acerca de las expresiones regulares en Capítulo 15.
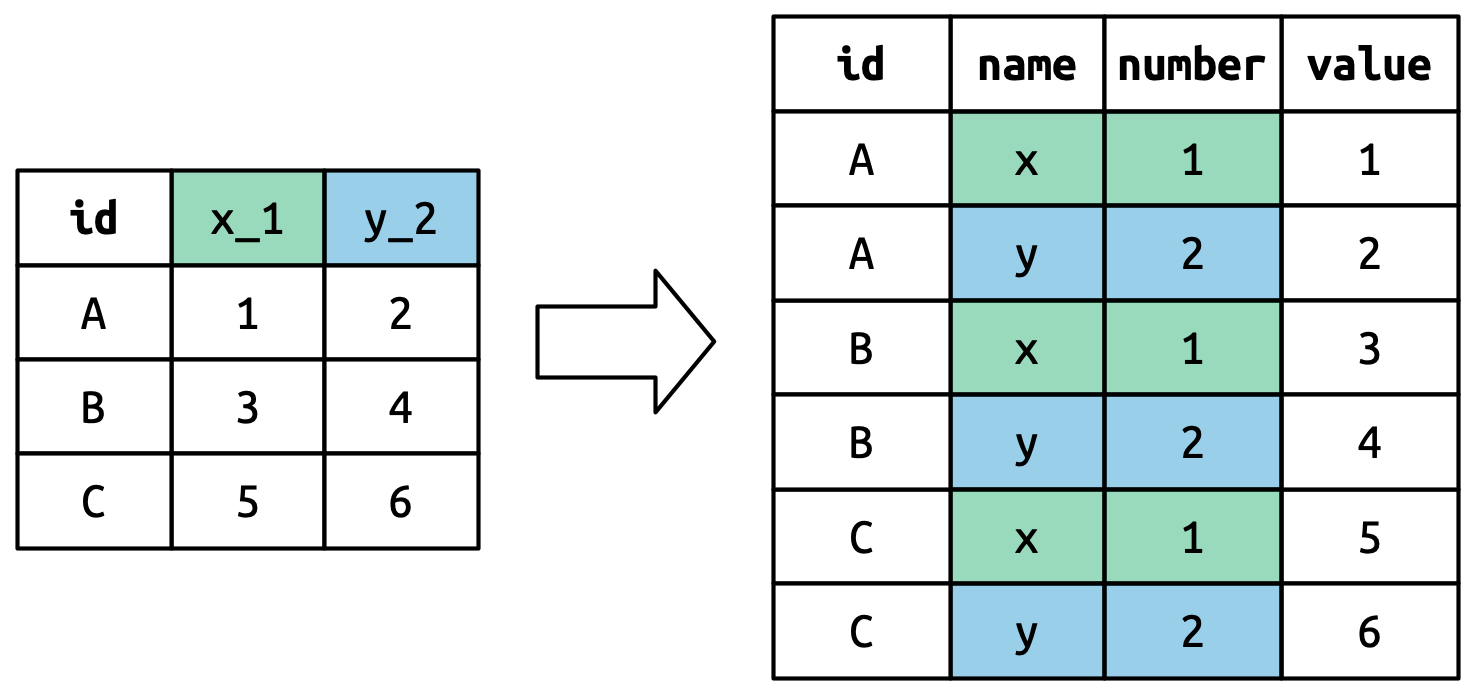
Conceptualmente, esta es solo una variación menor del caso más simple que ya ha visto. Figura 5.6 muestra la idea básica: ahora, en lugar de que los nombres de las columnas giren en una sola columna, giran en varias columnas. Puede imaginar que esto suceda en dos pasos (primero girando y luego separando), pero debajo del capó sucede en un solo paso porque eso es más rápido.
5.3.4 Datos y nombres de variables en los encabezados de las columnas
El siguiente paso en complejidad es cuando los nombres de las columnas incluyen una combinación de valores de variables y nombres de variables. Por ejemplo, tome el conjunto de datos household:
household
#> # A tibble: 5 × 5
#> family dob_child1 dob_child2 name_child1 name_child2
#> <int> <date> <date> <chr> <chr>
#> 1 1 1998-11-26 2000-01-29 Susan Jose
#> 2 2 1996-06-22 NA Mark <NA>
#> 3 3 2002-07-11 2004-04-05 Sam Seth
#> 4 4 2004-10-10 2009-08-27 Craig Khai
#> 5 5 2000-12-05 2005-02-28 Parker GracieEste conjunto de datos contiene información sobre cinco familias, con los nombres y fechas de nacimiento de hasta dos niños. El nuevo desafío en este conjunto de datos es que los nombres de las columnas contienen los nombres de dos variables (dob, name) y los valores de otra (child, con valores 1 o 2). Para resolver este problema, nuevamente necesitamos proporcionar un vector a names_to pero esta vez usamos el centinela especial ".value"; este no es el nombre de una variable sino un valor único que le dice a pivot_longer() que haga algo diferente. Esto anula el argumento values_to habitual para usar el primer componente del nombre de la columna dinámica como nombre de variable en la salida.
household |>
pivot_longer(
cols = !family,
names_to = c(".value", "child"),
names_sep = "_",
values_drop_na = TRUE
)
#> # A tibble: 9 × 4
#> family child dob name
#> <int> <chr> <date> <chr>
#> 1 1 child1 1998-11-26 Susan
#> 2 1 child2 2000-01-29 Jose
#> 3 2 child1 1996-06-22 Mark
#> 4 3 child1 2002-07-11 Sam
#> 5 3 child2 2004-04-05 Seth
#> 6 4 child1 2004-10-10 Craig
#> # ℹ 3 more rowsNuevamente usamos values_drop_na = TRUE, ya que la forma de la entrada fuerza la creación de variables faltantes explícitas (por ejemplo, para familias con un solo hijo).
Figura 5.7 ilustra la idea básica con un ejemplo más simple. Cuando usa ".value" en names_to, los nombres de las columnas en la entrada contribuyen tanto a los valores como a los nombres de las variables en la salida.
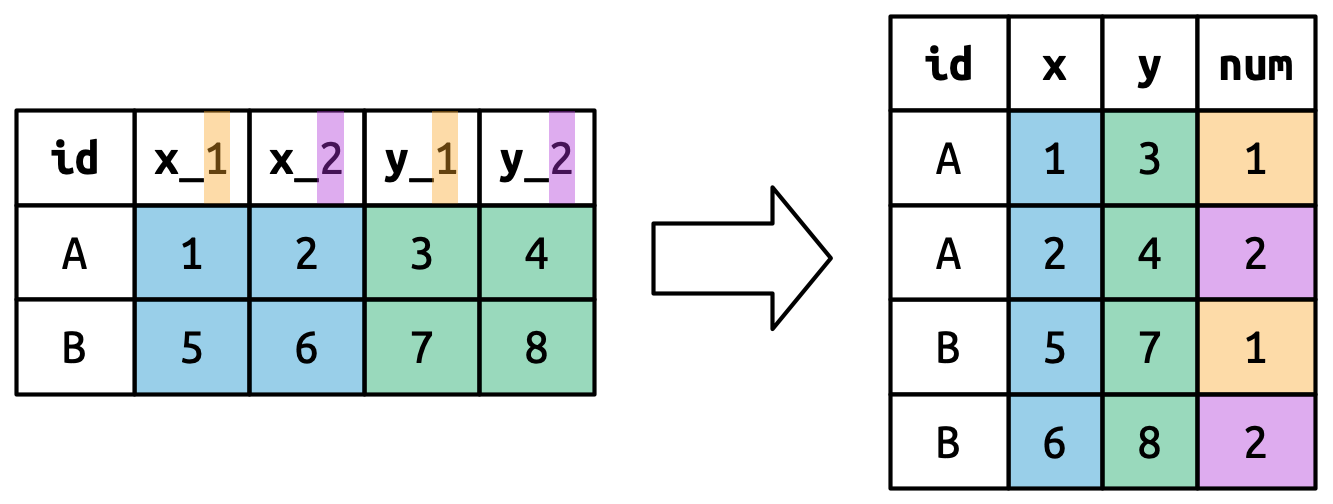
names_to = c(".value", "num") divide los nombres de las columnas en dos componentes: la primera parte determina la columna de salida nombre (x o y), y la segunda parte determina el valor de la columna num.
5.4 Ampliación de datos
Hasta ahora hemos usado pivot_longer() para resolver la clase común de problemas donde los valores terminan en los nombres de las columnas. A continuación, pivotaremos (HA HA) a pivot_wider(), que hace un conjunto de datos más ancho al incrementar el número de columnas y reducir las filas lo que ayuda cuando una observación se distribuye en varias filas. Esto parece surgir con menos frecuencia en la naturaleza, pero parece surgir mucho cuando se trata de datos gubernamentales.
Comenzaremos analizando cms_patient_experience, un conjunto de datos de los servicios de los Centros de Medicare y Medicaid que recopila datos sobre las experiencias de los pacientes:
cms_patient_experience
#> # A tibble: 500 × 5
#> org_pac_id org_nm measure_cd measure_title prf_rate
#> <chr> <chr> <chr> <chr> <dbl>
#> 1 0446157747 USC CARE MEDICAL GROUP INC CAHPS_GRP_1 CAHPS for MIPS… 63
#> 2 0446157747 USC CARE MEDICAL GROUP INC CAHPS_GRP_2 CAHPS for MIPS… 87
#> 3 0446157747 USC CARE MEDICAL GROUP INC CAHPS_GRP_3 CAHPS for MIPS… 86
#> 4 0446157747 USC CARE MEDICAL GROUP INC CAHPS_GRP_5 CAHPS for MIPS… 57
#> 5 0446157747 USC CARE MEDICAL GROUP INC CAHPS_GRP_8 CAHPS for MIPS… 85
#> 6 0446157747 USC CARE MEDICAL GROUP INC CAHPS_GRP_12 CAHPS for MIPS… 24
#> # ℹ 494 more rowsLa unidad central que se estudia es una organización, pero cada organización se distribuye en seis filas, con una fila para cada medición realizada en la organización de la encuesta. Podemos ver el conjunto completo de valores para measure_cd y measure_title usando distinct():
cms_patient_experience |>
distinct(measure_cd, measure_title)
#> # A tibble: 6 × 2
#> measure_cd measure_title
#> <chr> <chr>
#> 1 CAHPS_GRP_1 CAHPS for MIPS SSM: Getting Timely Care, Appointments, and In…
#> 2 CAHPS_GRP_2 CAHPS for MIPS SSM: How Well Providers Communicate
#> 3 CAHPS_GRP_3 CAHPS for MIPS SSM: Patient's Rating of Provider
#> 4 CAHPS_GRP_5 CAHPS for MIPS SSM: Health Promotion and Education
#> 5 CAHPS_GRP_8 CAHPS for MIPS SSM: Courteous and Helpful Office Staff
#> 6 CAHPS_GRP_12 CAHPS for MIPS SSM: Stewardship of Patient ResourcesNinguna de estas columnas hará nombres de variables particularmente buenos: measure_cd no sugiere el significado de la variable y measure_title es una oración larga que contiene espacios. Usaremos measure_cd como la fuente para nuestros nuevos nombres de columna por ahora, pero en un análisis real, es posible que desee crear sus propios nombres de variables que sean cortos y significativos.
`pivot_wider() tiene la interfaz opuesta a pivot_longer(): en lugar de elegir nuevos nombres de columna, debemos proporcionar las columnas existentes que definen los valores (values_from) y el nombre de la columna (names_from):
cms_patient_experience |>
pivot_wider(
names_from = measure_cd,
values_from = prf_rate
)
#> # A tibble: 500 × 9
#> org_pac_id org_nm measure_title CAHPS_GRP_1 CAHPS_GRP_2
#> <chr> <chr> <chr> <dbl> <dbl>
#> 1 0446157747 USC CARE MEDICAL GROUP … CAHPS for MIPS… 63 NA
#> 2 0446157747 USC CARE MEDICAL GROUP … CAHPS for MIPS… NA 87
#> 3 0446157747 USC CARE MEDICAL GROUP … CAHPS for MIPS… NA NA
#> 4 0446157747 USC CARE MEDICAL GROUP … CAHPS for MIPS… NA NA
#> 5 0446157747 USC CARE MEDICAL GROUP … CAHPS for MIPS… NA NA
#> 6 0446157747 USC CARE MEDICAL GROUP … CAHPS for MIPS… NA NA
#> # ℹ 494 more rows
#> # ℹ 4 more variables: CAHPS_GRP_3 <dbl>, CAHPS_GRP_5 <dbl>, …La salida no se ve muy bien; todavía parece que tenemos varias filas para cada organización. Eso es porque, también necesitamos decirle a pivot_wider() qué columna o columnas tienen valores que identifican de manera única cada fila; en este caso esas son las variables que comienzan con "org":
cms_patient_experience |>
pivot_wider(
id_cols = starts_with("org"),
names_from = measure_cd,
values_from = prf_rate
)
#> # A tibble: 95 × 8
#> org_pac_id org_nm CAHPS_GRP_1 CAHPS_GRP_2 CAHPS_GRP_3 CAHPS_GRP_5
#> <chr> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 0446157747 USC CARE MEDICA… 63 87 86 57
#> 2 0446162697 ASSOCIATION OF … 59 85 83 63
#> 3 0547164295 BEAVER MEDICAL … 49 NA 75 44
#> 4 0749333730 CAPE PHYSICIANS… 67 84 85 65
#> 5 0840104360 ALLIANCE PHYSIC… 66 87 87 64
#> 6 0840109864 REX HOSPITAL INC 73 87 84 67
#> # ℹ 89 more rows
#> # ℹ 2 more variables: CAHPS_GRP_8 <dbl>, CAHPS_GRP_12 <dbl>Esto nos da la salida que estamos buscando.
5.4.1 ¿Cómo funciona pivot_wider()?
Para entender cómo funciona pivot_wider(), comencemos nuevamente con un conjunto de datos muy simple. Esta vez tenemos dos pacientes con ids A y B, tenemos tres mediciones de presión arterial en el paciente A y dos en el paciente B:
df <- tribble(
~id, ~measurement, ~value,
"A", "bp1", 100,
"B", "bp1", 140,
"B", "bp2", 115,
"A", "bp2", 120,
"A", "bp3", 105
)Tomaremos los valores de la columna value y los nombres de la columna measurement:
df |>
pivot_wider(
names_from = measurement,
values_from = value
)
#> # A tibble: 2 × 4
#> id bp1 bp2 bp3
#> <chr> <dbl> <dbl> <dbl>
#> 1 A 100 120 105
#> 2 B 140 115 NAPara comenzar el proceso, pivot_wider() necesita primero averiguar qué irá en las filas y columnas. Los nuevos nombres de columna serán los valores únicos de measurement.
De forma predeterminada, las filas de la salida están determinadas por todas las variables que no se incluirán en los nuevos nombres o valores. Estos se llaman id_cols. Aquí solo hay una columna, pero en general puede haber cualquier cantidad de columnas.
pivot_wider() luego combina estos resultados para generar un data frame vacío:
Luego completa todos los valores faltantes usando los datos en la entrada. En este caso, no todas las celdas de la salida tienen un valor correspondiente en la entrada, ya que no hay una tercera medición de la presión arterial para el paciente B, por lo que falta esa celda. Volveremos a esta idea de que pivot_wider() puede “hacer” valores faltantes en Capítulo 18.
También puede preguntarse qué sucede si hay varias filas en la entrada que corresponden a una celda en la salida. El siguiente ejemplo tiene dos filas que corresponden a el id “A” y a measurement “bp1”:
df <- tribble(
~id, ~measurement, ~value,
"A", "bp1", 100,
"A", "bp1", 102,
"A", "bp2", 120,
"B", "bp1", 140,
"B", "bp2", 115
)Si intentamos pivotar esto, obtenemos una salida que contiene columnas de lista, sobre las que aprenderá más en Capítulo 23:
df |>
pivot_wider(
names_from = measurement,
values_from = value
)
#> Warning: Values from `value` are not uniquely identified; output will contain
#> list-cols.
#> • Use `values_fn = list` to suppress this warning.
#> • Use `values_fn = {summary_fun}` to summarise duplicates.
#> • Use the following dplyr code to identify duplicates.
#> {data} |>
#> dplyr::summarise(n = dplyr::n(), .by = c(id, measurement)) |>
#> dplyr::filter(n > 1L)
#> # A tibble: 2 × 3
#> id bp1 bp2
#> <chr> <list> <list>
#> 1 A <dbl [2]> <dbl [1]>
#> 2 B <dbl [1]> <dbl [1]>Como aún no sabe cómo trabajar con este tipo de datos, querrá seguir la sugerencia de la advertencia para averiguar dónde está el problema:
Luego, depende de usted averiguar qué salió mal con sus datos y reparar el daño subyacente o usar sus habilidades de agrupación y resumen para asegurarse de que cada combinación de valores de fila y columna solo tenga una fila.
5.5 Resumen
En este capítulo aprendiste sobre datos ordenados: datos que tienen variables en columnas y observaciones en filas. Los datos ordenados facilitan el trabajo en el tidyverse, porque es una estructura consistente que la mayoría de las funciones entienden, el principal desafío es transformar los datos de cualquier estructura en la que los reciba a un formato ordenado. Con ese fin, aprendió sobre pivot_longer() y pivot_wider(), que le permiten ordenar muchos conjuntos de datos desordenados. Los ejemplos que presentamos aquí son una selección de los de vignette("pivot", package = "tidyr"), por lo que si encuentra un problema con el que este capítulo no le ayuda, esa viñeta es un buen lugar para probar próximo.
Otro desafío es que, para un conjunto de datos dado, puede ser imposible etiquetar la versión más larga o más amplia como la “ordenada”. Esto es en parte un reflejo de nuestra definición de datos ordenados, donde dijimos que los datos ordenados tienen una variable en cada columna, pero en realidad no definimos qué es una variable (y es sorprendentemente difícil hacerlo). Está totalmente bien ser pragmático y decir que una variable es lo que hace que su análisis sea más fácil. Entonces, si no sabe cómo hacer algunos cálculos, considere cambiar la organización de sus datos; ¡no tenga miedo de desordenar, transformar y volver a ordenar según sea necesario!
Si disfrutó de este capítulo y desea obtener más información sobre la teoría subyacente, puede obtener más información sobre la historia y los fundamentos teóricos en el artículo Tidy Data publicado. en el Journal of Statistical Software.
Ahora que está escribiendo una cantidad sustancial de código R, es hora de aprender más sobre cómo organizar su código en archivos y directorios. En el próximo capítulo, aprenderá todo acerca de las ventajas de los scripts y proyectos, y algunas de las muchas herramientas que brindan para facilitarle la vida.
La canción se incluirá siempre que haya estado entre las 100 mejores en algún momento del año 2000, y se rastreará hasta 72 semanas después de su aparición.↩︎
Volveremos sobre esta idea en Capítulo 18.↩︎