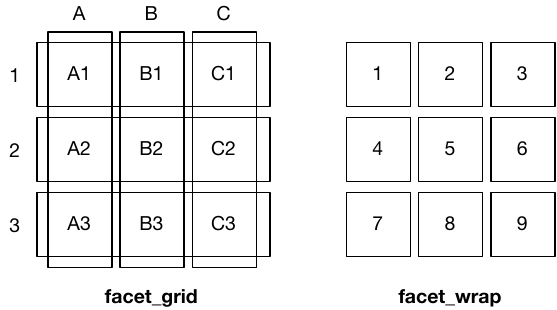
facet_grid() (izquierda) es fundamentalmente 2d y se compone de dos componentes independientes. facet_wrap() (derecha) es 1d, pero está envuelto en 2d para ahorrar espacio.You are reading the work-in-progress third edition of the ggplot2 book. This chapter is currently a dumping ground for ideas, and we don’t recommend reading it.
La primera vez que encontró facetado fue en Sección 2.5. El facetado genera pequeños múltiplos, cada uno de los cuales muestra un subconjunto diferente de datos. Los múltiplos pequeños son una herramienta poderosa para el análisis exploratorio de datos: puede comparar rápidamente patrones en diferentes partes de los datos y ver si son iguales o diferentes. Esta sección discutirá cómo puede ajustar las facetas, particularmente la forma en que interactúan con las escalas de posición.
Hay tres tipos de facetado:
facet_null(): una sola gráfica, el valor predeterminado.
facet_wrap(): “agrupa” una cinta 1d de paneles en 2d.
facet_grid(): produce una cuadrícula 2D de paneles definidos por variables que forman las filas y columnas.
Las diferencias entre facet_wrap() y facet_grid() se ilustran en la siguiente figura.

facet_grid() (izquierda) es fundamentalmente 2d y se compone de dos componentes independientes. facet_wrap() (derecha) es 1d, pero está envuelto en 2d para ahorrar espacio.Los gráficos facetados tienen la capacidad de llenar una gran cantidad de espacio, por lo que para este capítulo usaremos un subconjunto del conjunto de datos de mpg que tiene una cantidad manejable de niveles: tres cilindros (4, 6, 8), dos tipos de tren motriz ( 4 y f), y seis clases.
facet_wrap() crea una cinta larga de paneles (generada por cualquier número de variables) y la envuelve en 2d. Esto es útil si tiene una única variable con muchos niveles y desea organizar los gráficos de una manera más eficiente en cuanto a espacio.
Puede controlar cómo se envuelve la cinta en una cuadrícula con ncol, nrow, as.table y dir. ncol y nrow controlan cuántas columnas y filas (solo necesita configurar una). as.table controla si las facetas se presentan como una tabla (TRUE), con los valores más altos en la parte inferior derecha, o como un gráfico (FALSE), con los valores más altos en la parte superior derecha. dir controla la dirección de ajuste: horizontal o vertical.
base <- ggplot(mpg2, aes(displ, hwy)) +
geom_blank() +
xlab(NULL) +
ylab(NULL)
base + facet_wrap(~class, ncol = 3)
base + facet_wrap(~class, ncol = 3, as.table = FALSE)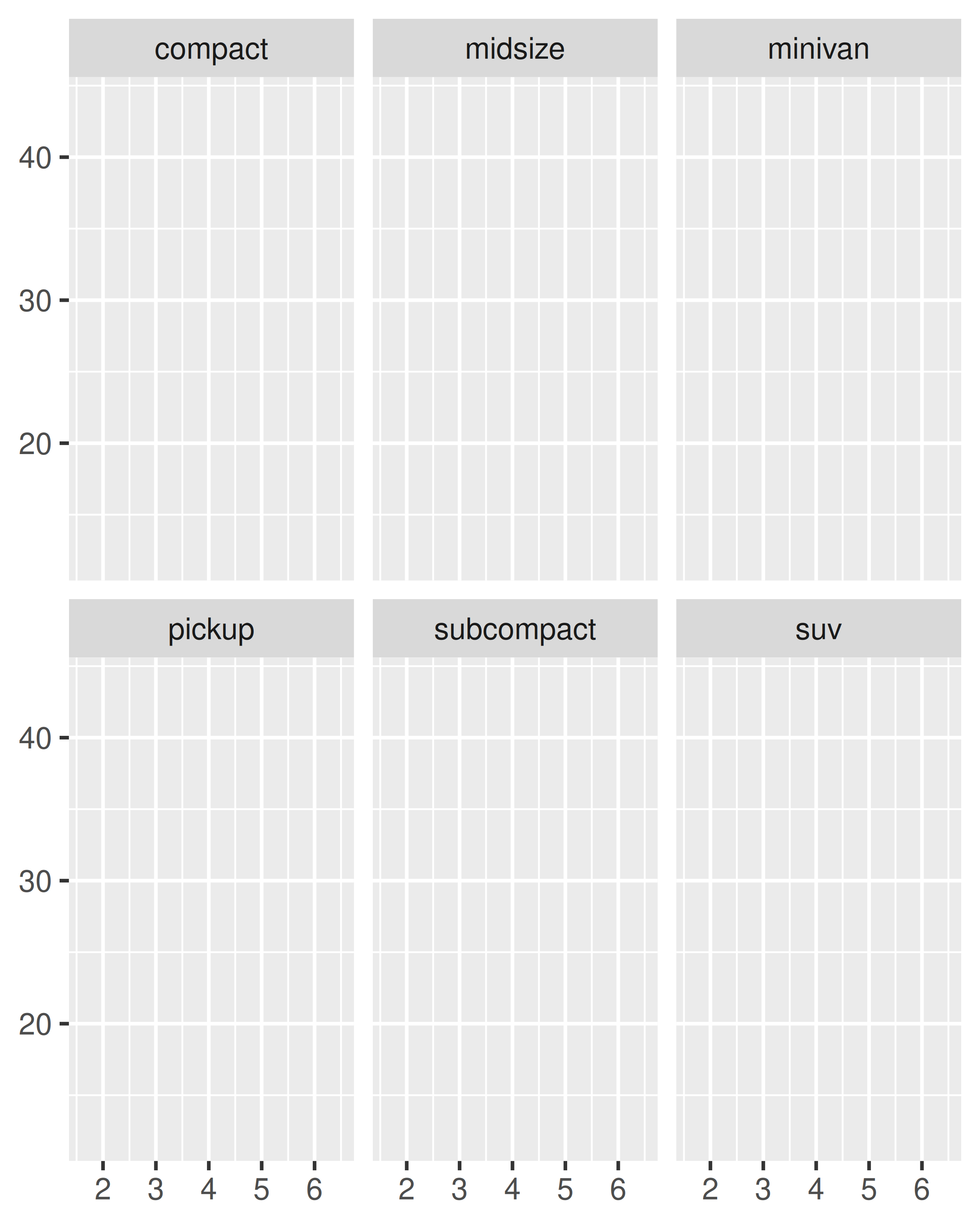
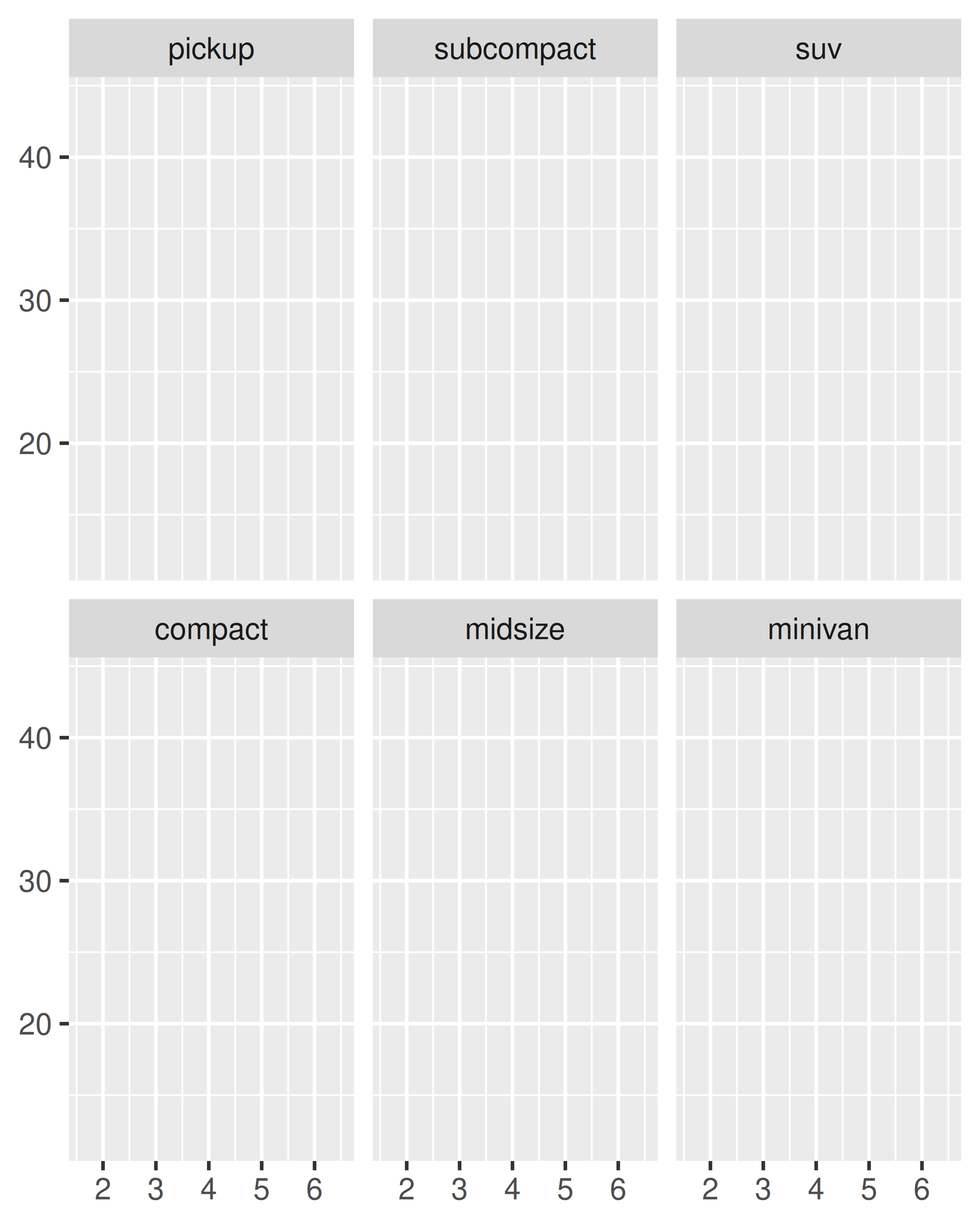
base + facet_wrap(~class, nrow = 3)
base + facet_wrap(~class, nrow = 3, dir = "v")
facet_grid() presenta gráficos en una cuadrícula 2D, según lo definido por una fórmula:
. ~ a distribuye los valores de a entre las columnas. Esta dirección facilita las comparaciones de la posición y, porque las escalas verticales están alineadas.
base + facet_grid(. ~ cyl)
b ~ . distribuye los valores de b por las filas. Esta dirección facilita la comparación de la posición x porque las escalas horizontales están alineadas. Esto lo hace particularmente útil para comparar distribuciones.
base + facet_grid(drv ~ .)
b ~ a distribuye a en las columnas y b en las filas. Por lo general, querrás colocar la variable con la mayor cantidad de niveles en las columnas para aprovechar la relación de aspecto de tu pantalla.
base + facet_grid(drv ~ cyl)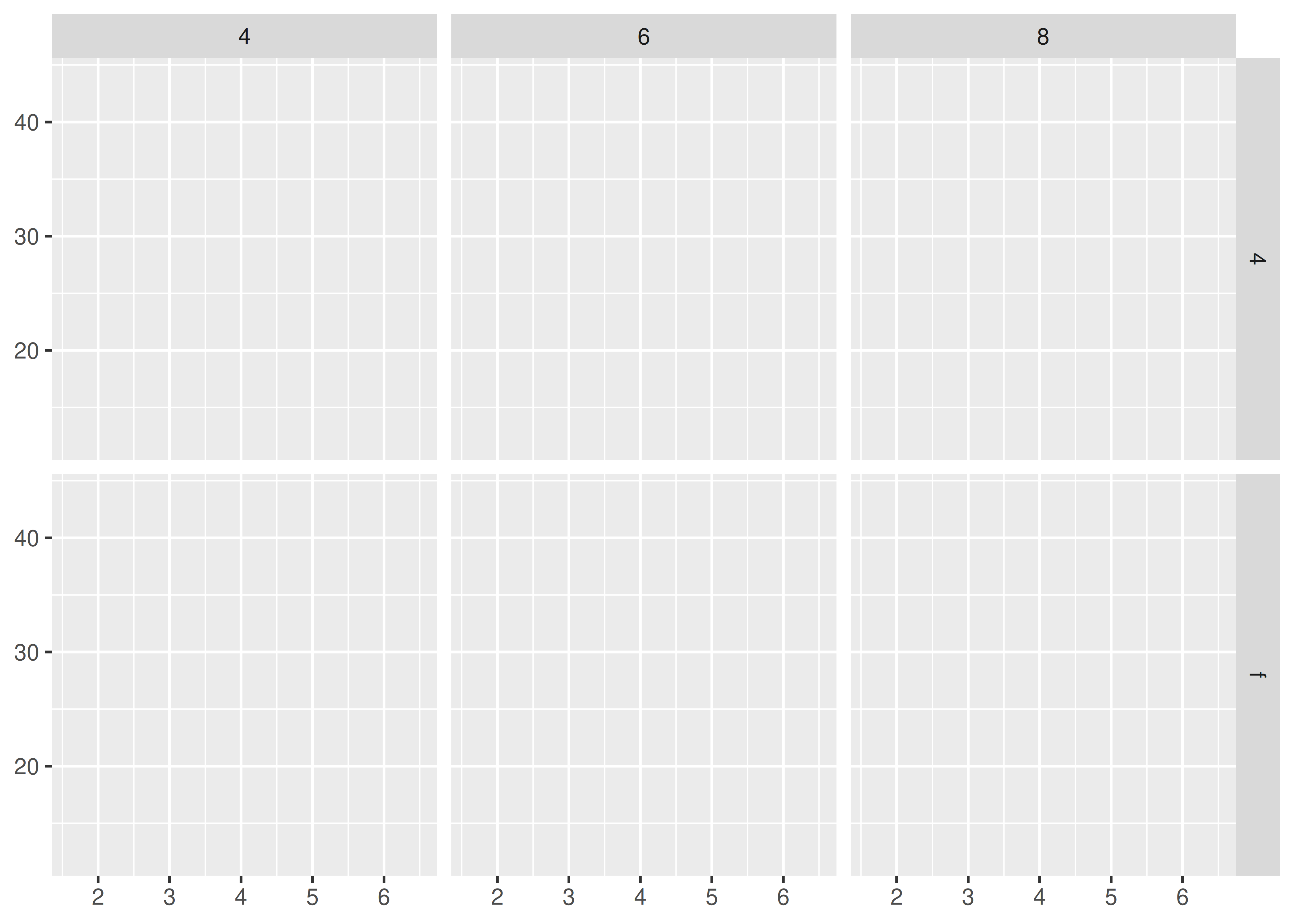
Puede utilizar varias variables en las filas o columnas, “sumándolas”, p. a + b ~ c + d. Las variables que aparecen juntas en las filas o columnas están anidadas en el sentido de que solo las combinaciones que aparecen en los datos aparecerán en el gráfico. Las variables que se especifican en filas y columnas se cruzarán: se mostrarán todas las combinaciones, incluidas aquellas que no aparecieron en el conjunto de datos original: esto puede dar como resultado paneles vacíos.
Tanto para facet_wrap() como para facet_grid() puedes controlar si las escalas de posición son las mismas en todos los paneles (fijas) o si se les permite variar entre paneles (gratis) con el parámetro scales:
scales = "fixed": las escalas x e y están fijadas en todos los paneles.scales = "free_x": la escala x es libre y la escala y es fija.scales = "free_y": la escala y es libre y la escala x es fija.scales = "free": las escalas x e y varían según los paneles.facet_grid() impone una restricción adicional a las escalas: todos los paneles de una columna deben tener la misma escala x y todos los paneles de una fila deben tener la misma escala y. Esto se debe a que cada columna comparte un eje x y cada fila comparte un eje y.
Las escalas fijas facilitan la visualización de patrones entre paneles; Las escalas libres facilitan la visualización de patrones dentro de los paneles.
p <- ggplot(mpg2, aes(cty, hwy)) +
geom_abline() +
geom_jitter(width = 0.1, height = 0.1)
p + facet_wrap(~cyl)
p + facet_wrap(~cyl, scales = "free")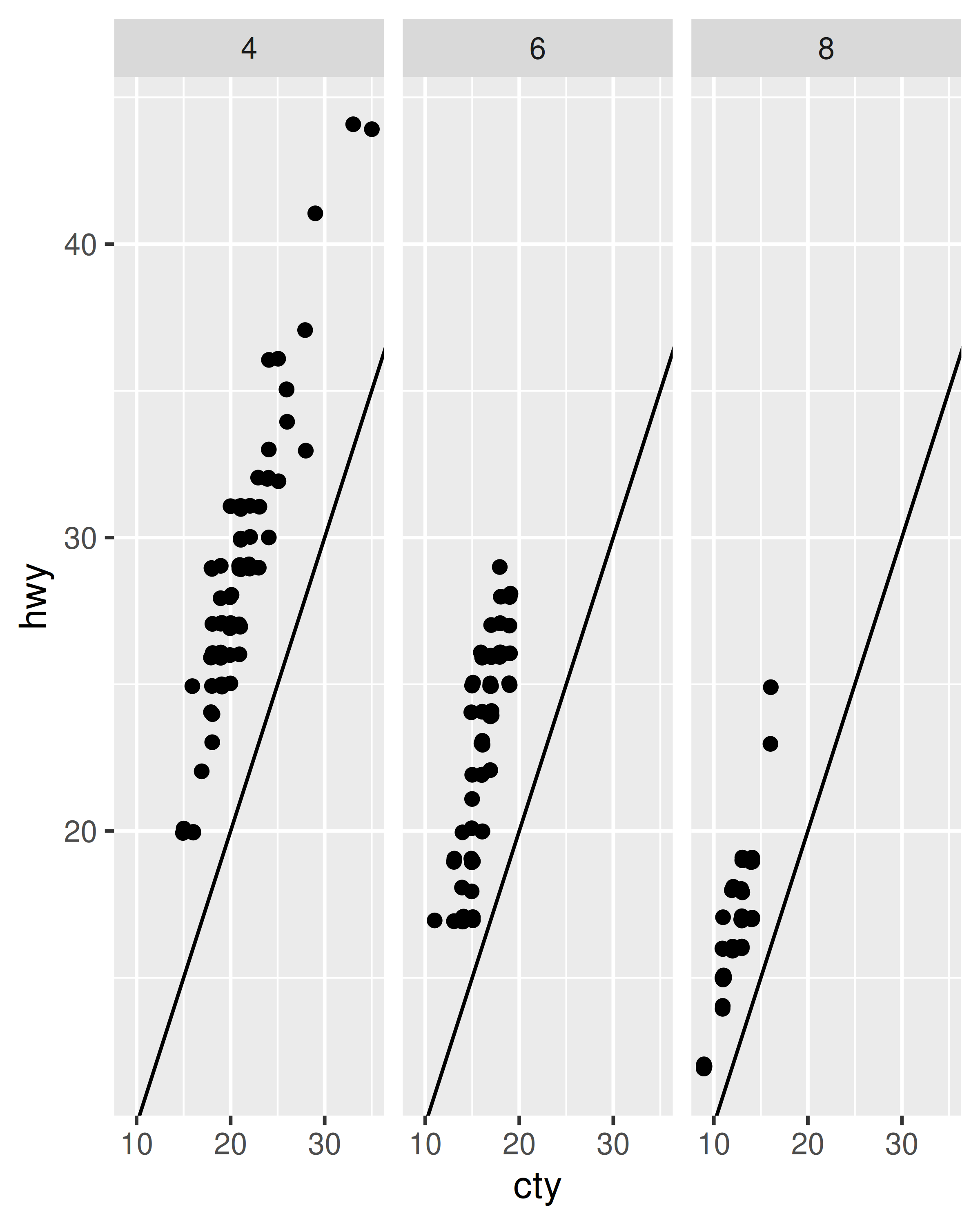
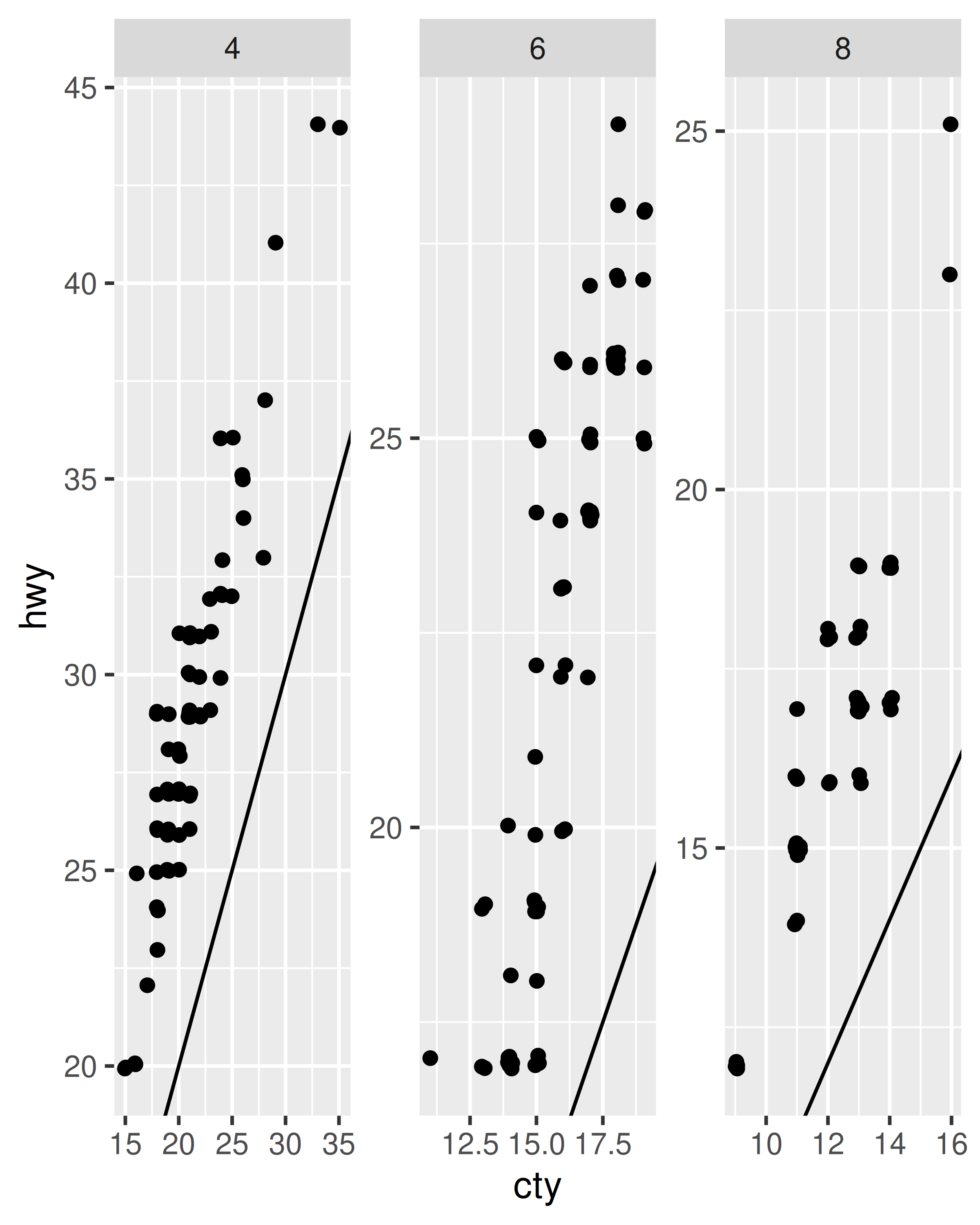
Las escalas libres también son útiles cuando queremos mostrar múltiples series de tiempo medidas en diferentes escalas. Para hacer esto, primero necesitamos cambiar de datos “anchos” a “largos”, apilando las variables separadas en una sola columna. A continuación se muestra un ejemplo de esto con la forma larga de los datos economics.
economics_long
#> # A tibble: 2,870 × 4
#> date variable value value01
#> <date> <chr> <dbl> <dbl>
#> 1 1967-07-01 pce 507. 0
#> 2 1967-08-01 pce 510. 0.000265
#> 3 1967-09-01 pce 516. 0.000762
#> 4 1967-10-01 pce 512. 0.000471
#> 5 1967-11-01 pce 517. 0.000916
#> 6 1967-12-01 pce 525. 0.00157
#> # ℹ 2,864 more rows
ggplot(economics_long, aes(date, value)) +
geom_line() +
facet_wrap(~variable, scales = "free_y", ncol = 1)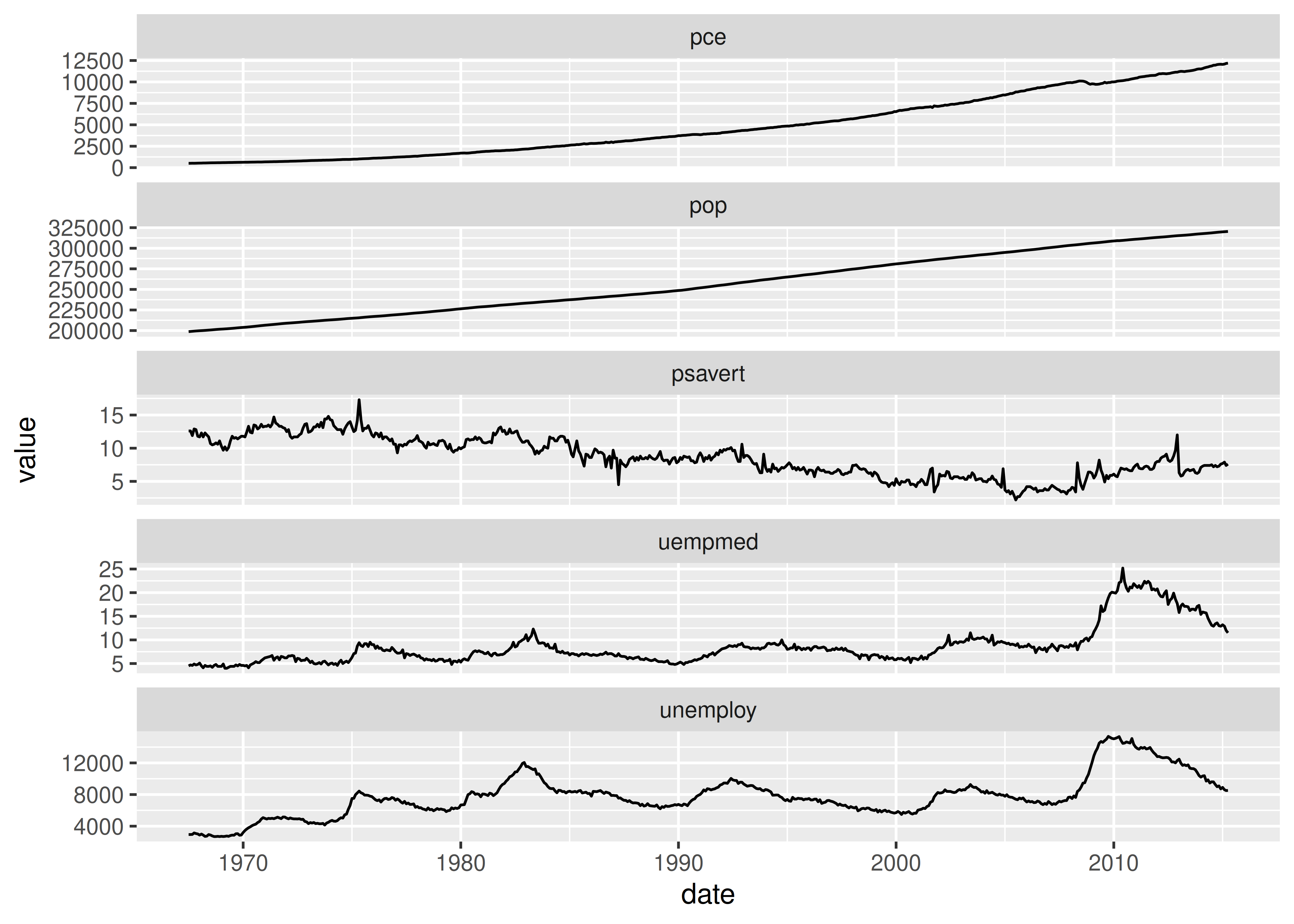
facet_grid() tiene un parámetro adicional llamado space, que toma los mismos valores que scales. Cuando el espacio está “free”, cada columna (o fila) tendrá un ancho (o alto) proporcional al rango de la escala para esa columna (o fila). Esto hace que la escala sea igual en todo el gráfico: 1 cm en cada panel se asigna al mismo rango de datos. (Esto es algo análogo a los límites del eje ‘cortado’ de la red). Por ejemplo, si el panel a tuviera un rango 2 y el panel b tuviera un rango 4, un tercio del espacio se le daría a a y dos tercios a b. . Esto es más útil para escalas categóricas, donde podemos asignar espacio proporcionalmente en función del número de niveles en cada faceta, como se ilustra a continuación.
Si está utilizando facetado en un gráfico con múltiples conjuntos de datos, ¿qué sucede cuando a uno de esos conjuntos de datos le faltan las variables de facetado? Esta situación surge comúnmente cuando agrega información contextual que debería ser la misma en todos los paneles. Por ejemplo, imagine que tiene una visualización espacial de una enfermedad dividida por género. ¿Qué sucede cuando agrega una capa de mapa que no contiene la variable de género? Aquí ggplot hará lo que espera: mostrará el mapa en cada faceta: las variables de facetas faltantes se tratan como si tuvieran todos los valores.
He aquí un ejemplo sencillo. Observe cómo aparece el único punto rojo de df2 en ambos paneles.
df1 <- data.frame(x = 1:3, y = 1:3, gender = c("f", "f", "m"))
df2 <- data.frame(x = 2, y = 2)
ggplot(df1, aes(x, y)) +
geom_point(data = df2, colour = "red", size = 2) +
geom_point() +
facet_wrap(~gender)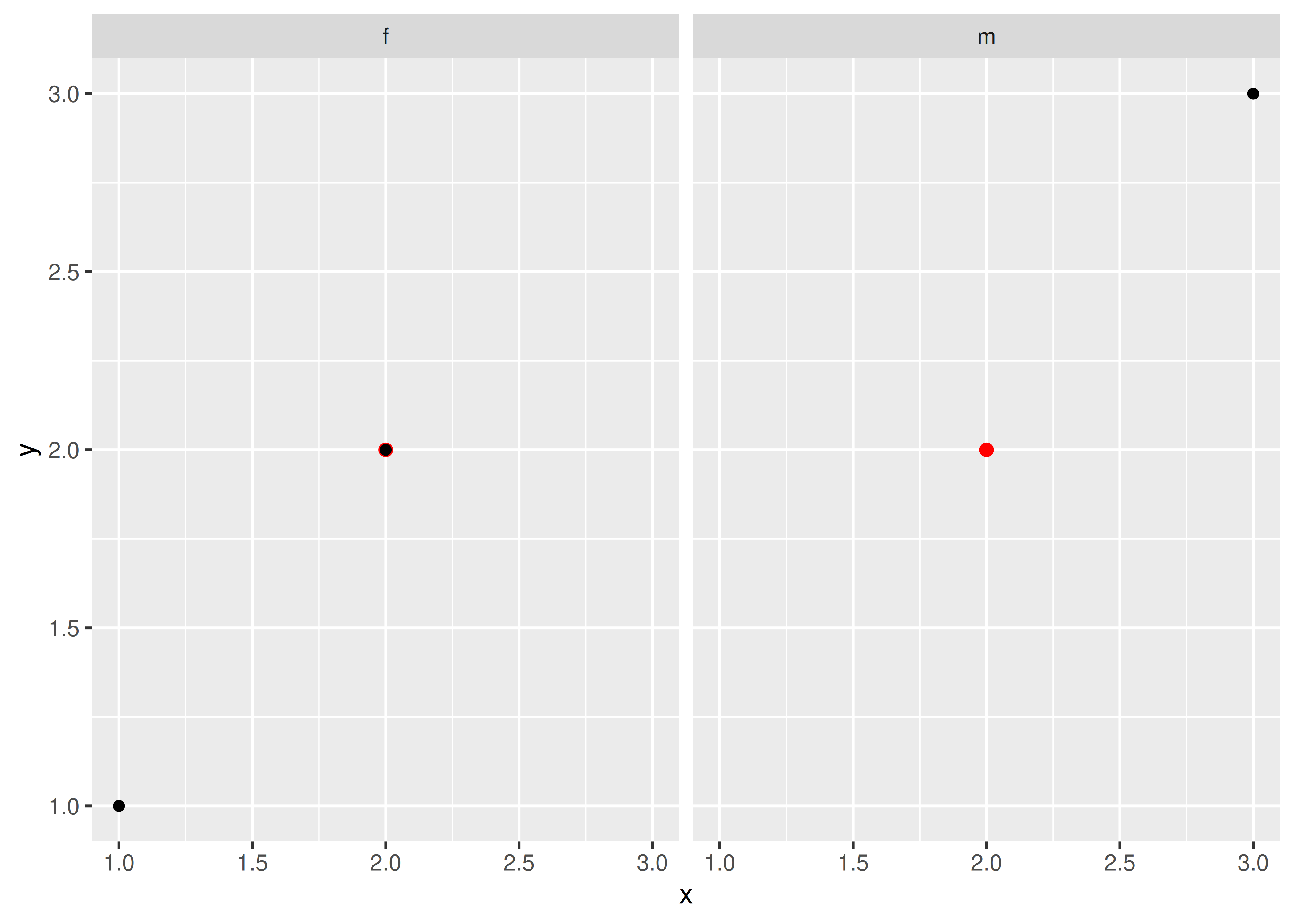
Esta técnica es particularmente útil cuando agrega anotaciones para facilitar la comparación entre facetas, como se muestra en la siguiente sección.
El facetado es una alternativa al uso de la estética (como el color, la forma o el tamaño) para diferenciar grupos. Ambas técnicas tienen fortalezas y debilidades, basadas en las posiciones relativas de los subconjuntos. Con el facetado, cada grupo está bastante alejado en su propio panel y no hay superposición entre los grupos. Esto es bueno si los grupos se superponen mucho, pero hace que las pequeñas diferencias sean más difíciles de ver. Cuando se utiliza la estética para diferenciar grupos, los grupos están muy juntos y pueden superponerse, pero las pequeñas diferencias son más fáciles de ver.
df <- data.frame(
x = rnorm(120, c(0, 2, 4)),
y = rnorm(120, c(1, 2, 1)),
z = letters[1:3]
)
ggplot(df, aes(x, y)) +
geom_point(aes(colour = z))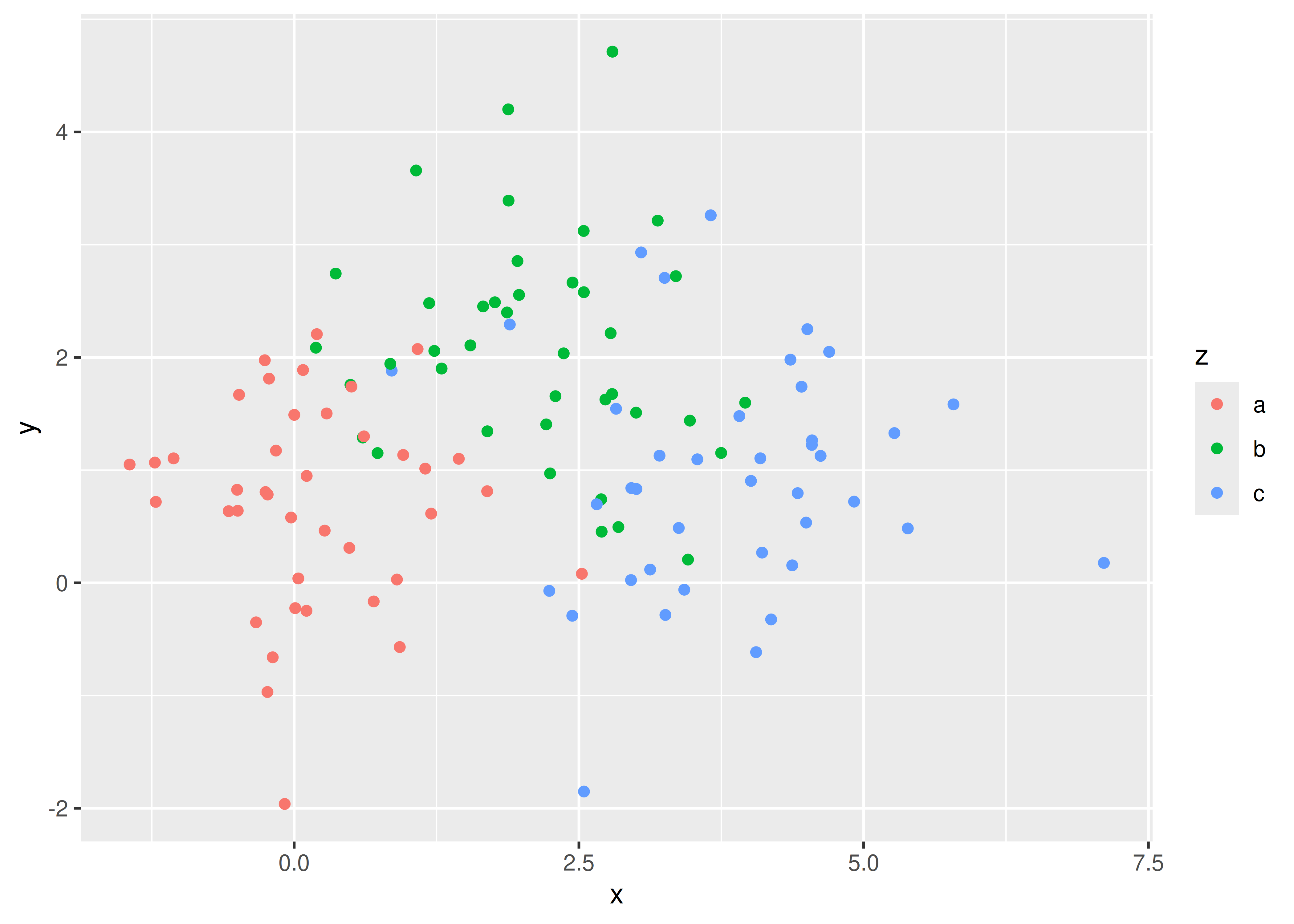
ggplot(df, aes(x, y)) +
geom_point() +
facet_wrap(~z)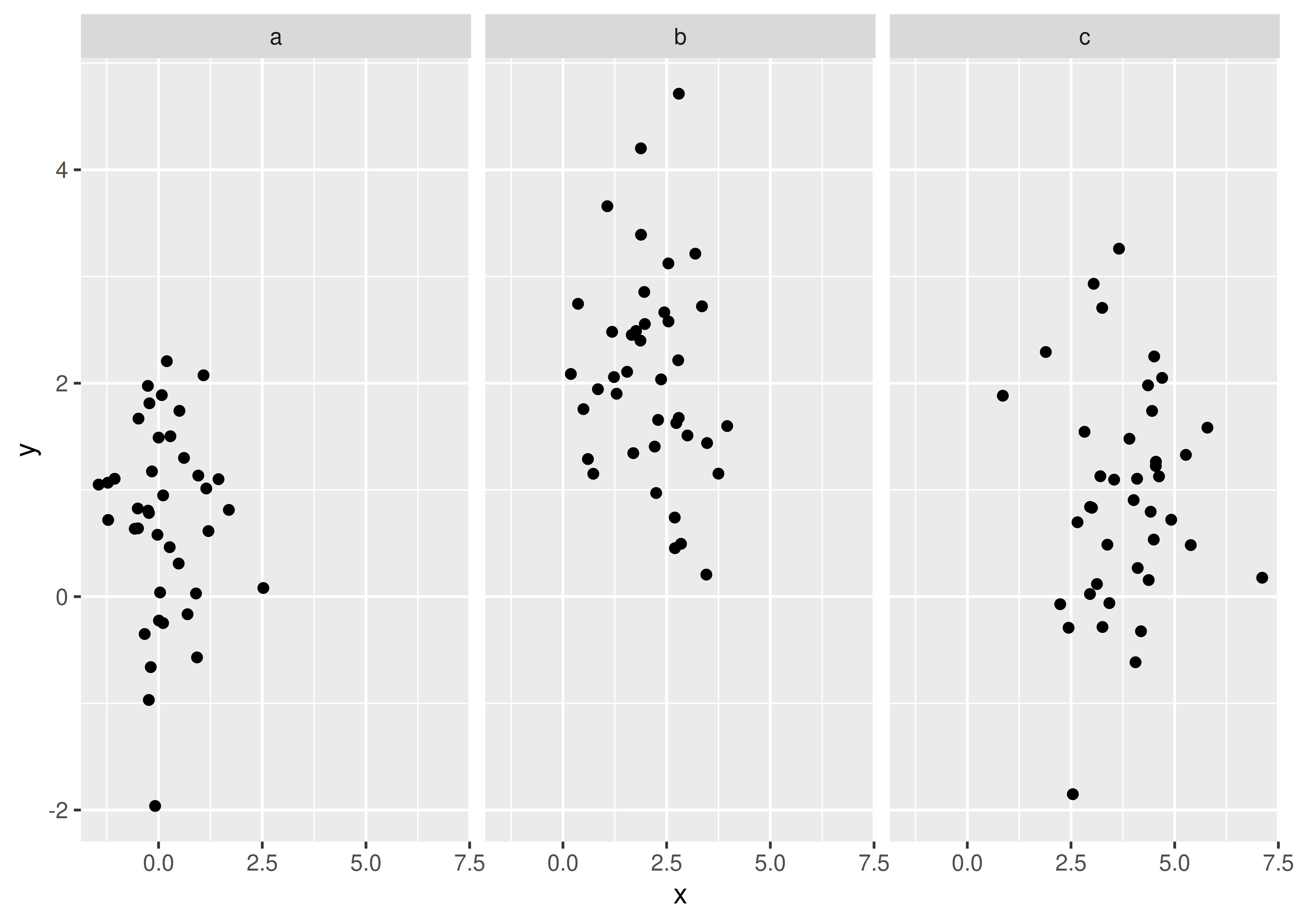
Las comparaciones entre facetas a menudo se benefician de alguna anotación reflexiva. Por ejemplo, en este caso podríamos mostrar la media de cada grupo en cada panel. Para ello, agrupamos y resumimos los datos utilizando el paquete dplyr, que se trata en R para ciencia de datos en https://r4ds.had.co.nz. Tenga en cuenta que necesitamos dos variables “z”: una para las facetas y otra para los colores.
df_sum <- df %>%
group_by(z) %>%
summarise(x = mean(x), y = mean(y)) %>%
rename(z2 = z)
ggplot(df, aes(x, y)) +
geom_point() +
geom_point(data = df_sum, aes(colour = z2), size = 4) +
facet_wrap(~z)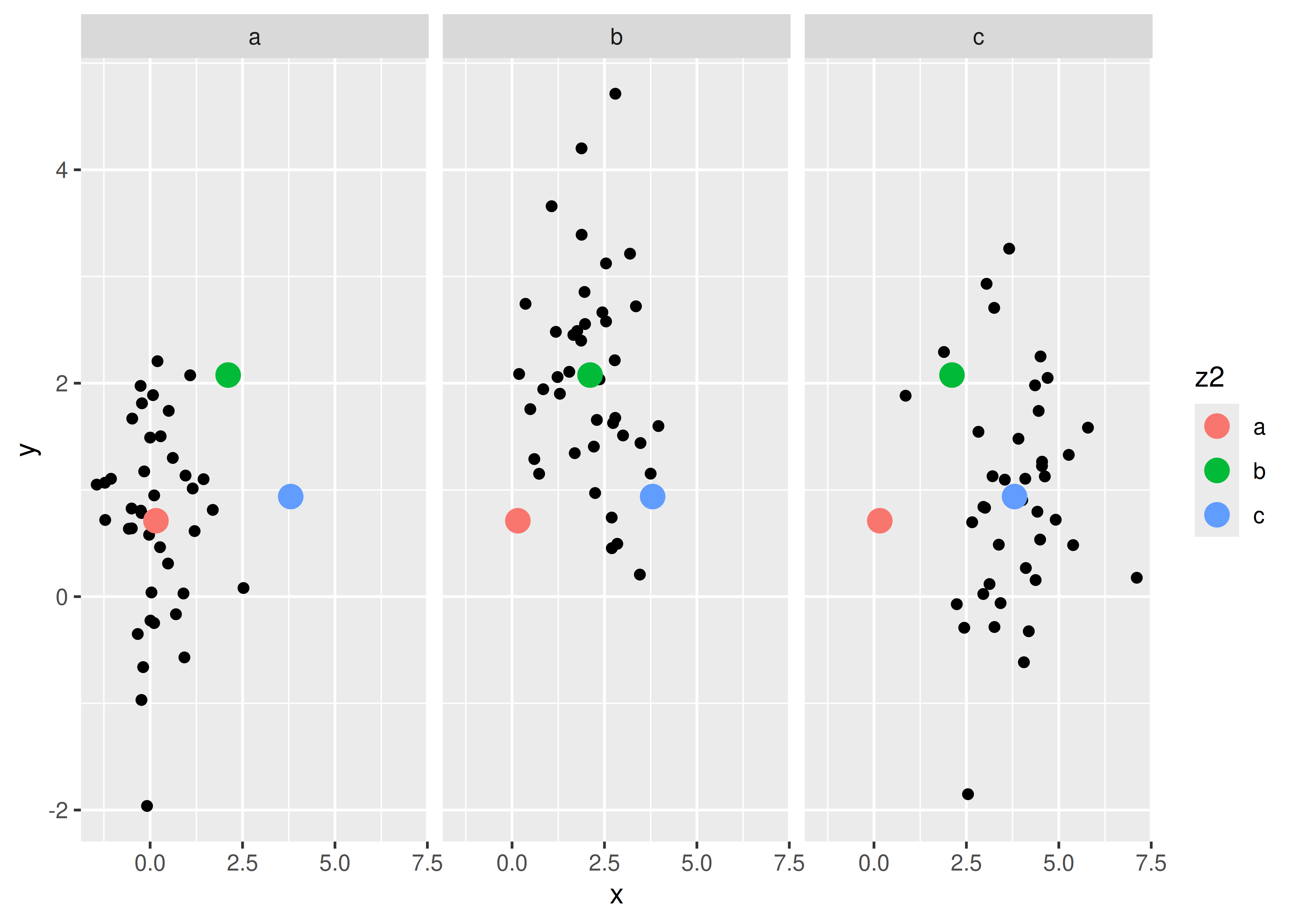
Otra técnica útil es poner todos los datos en el fondo de cada panel:
df2 <- dplyr::select(df, -z)
ggplot(df, aes(x, y)) +
geom_point(data = df2, colour = "grey70") +
geom_point(aes(colour = z)) +
facet_wrap(~z)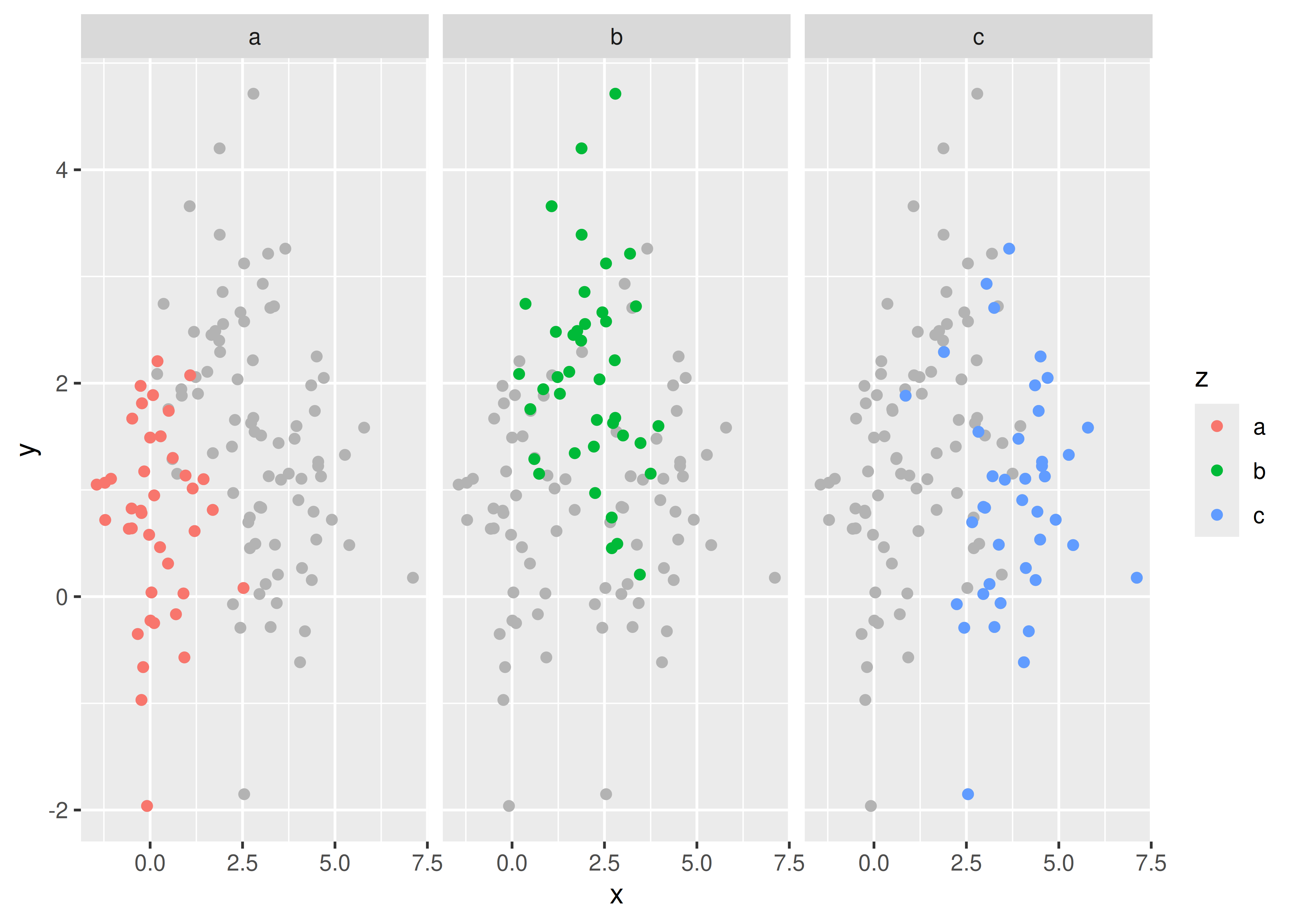
Para facetar variables continuas, primero debe discretizarlas. ggplot2 proporciona tres funciones auxiliares para hacerlo:
Divida los datos en n contenedores, cada uno de ellos de la misma longitud: cut_interval(x, n)
Divida los datos en contenedores de ancho width: cut_width(x, width).
Divida los datos en n contenedores, cada uno de los cuales contiene (aproximadamente) la misma cantidad de puntos: cut_number(x, n = 10).
Se ilustran a continuación:
# Contenedores de ancho 1
mpg2$disp_w <- cut_width(mpg2$displ, 1)
# Seis contenedores de igual longitud
mpg2$disp_i <- cut_interval(mpg2$displ, 6)
# Seis contenedores que contienen el mismo número de puntos
mpg2$disp_n <- cut_number(mpg2$displ, 6)
plot <- ggplot(mpg2, aes(cty, hwy)) +
geom_point() +
labs(x = NULL, y = NULL)
plot + facet_wrap(~disp_w, nrow = 1)
plot + facet_wrap(~disp_i, nrow = 1)
plot + facet_wrap(~disp_n, nrow = 1)
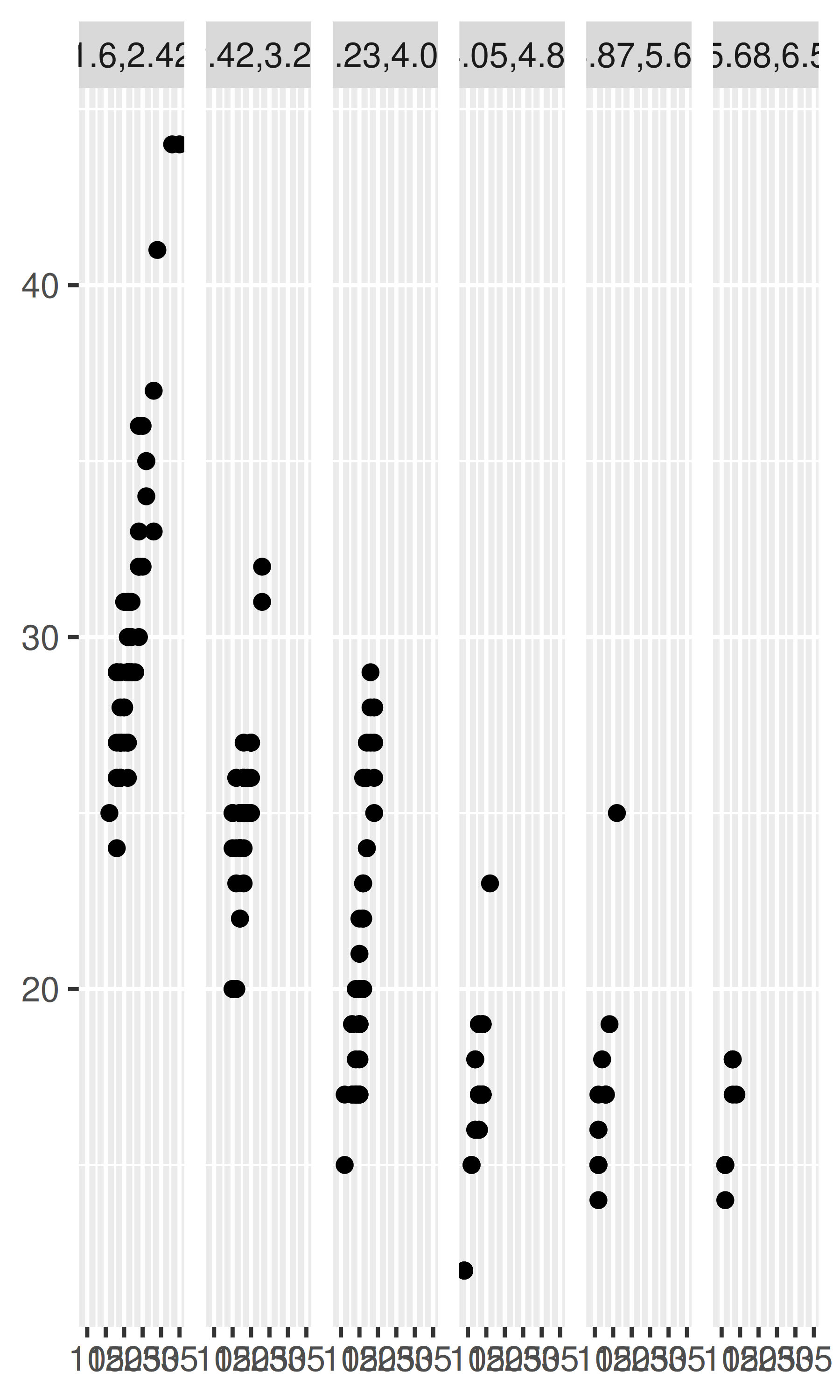
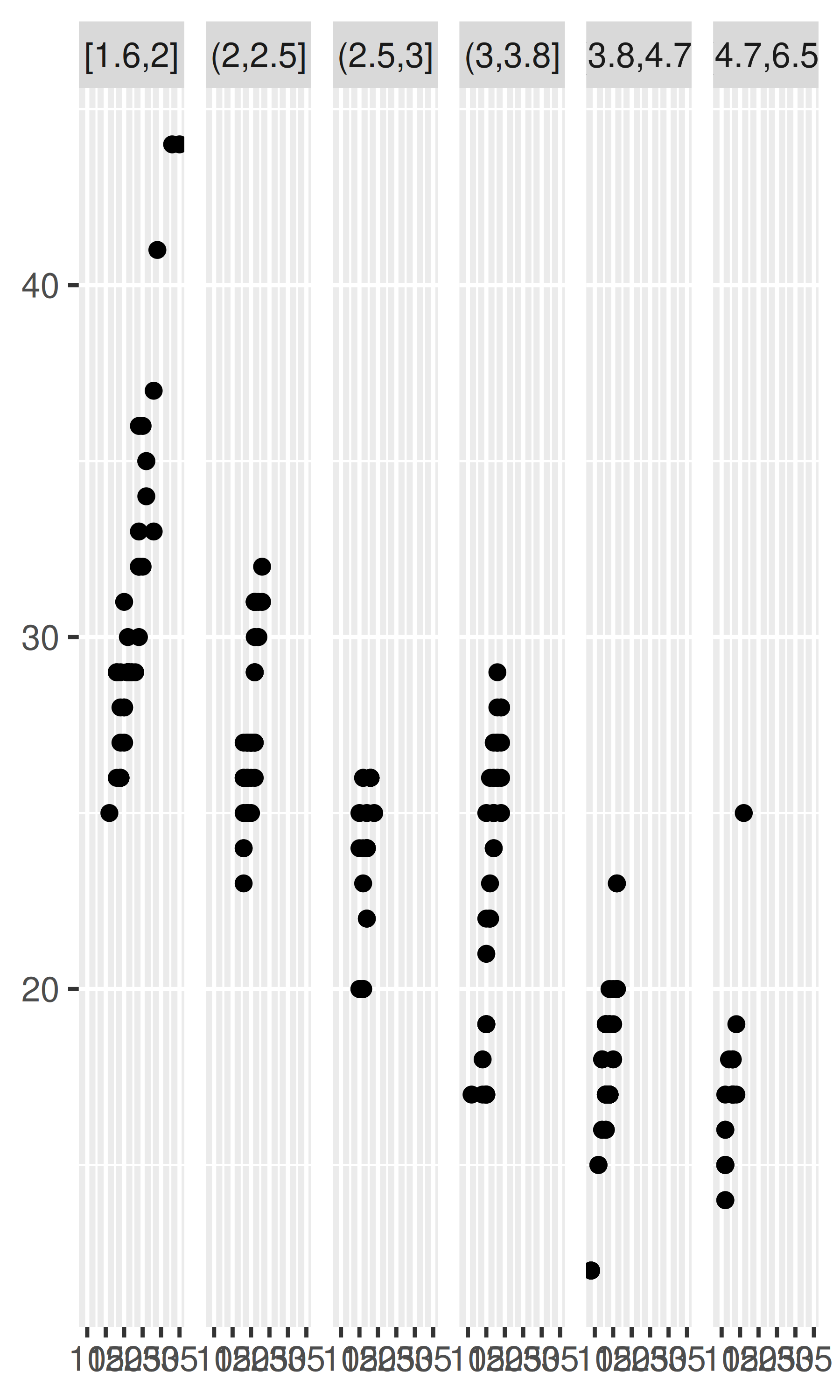
Tenga en cuenta que la fórmula de facetado no evalúa funciones, por lo que primero debe crear una nueva variable que contenga los datos discretizados.
Diamantes: muestra la distribución del precio condicionada a la talla y los quilates. Intente facetar por corte y agrupar por quilates. Intente facetar por quilates y agrupar por corte. ¿Cual prefieres?
Diamantes: compara la relación entre precio y quilates de cada color. ¿Qué hace que sea difícil comparar los grupos? ¿Es mejor agrupar o facetar? Si utiliza facetado, ¿qué anotación podría agregar para que sea más fácil ver las diferencias entre los paneles?
¿Por qué facet_wrap() generalmente es más útil que facet_grid()?
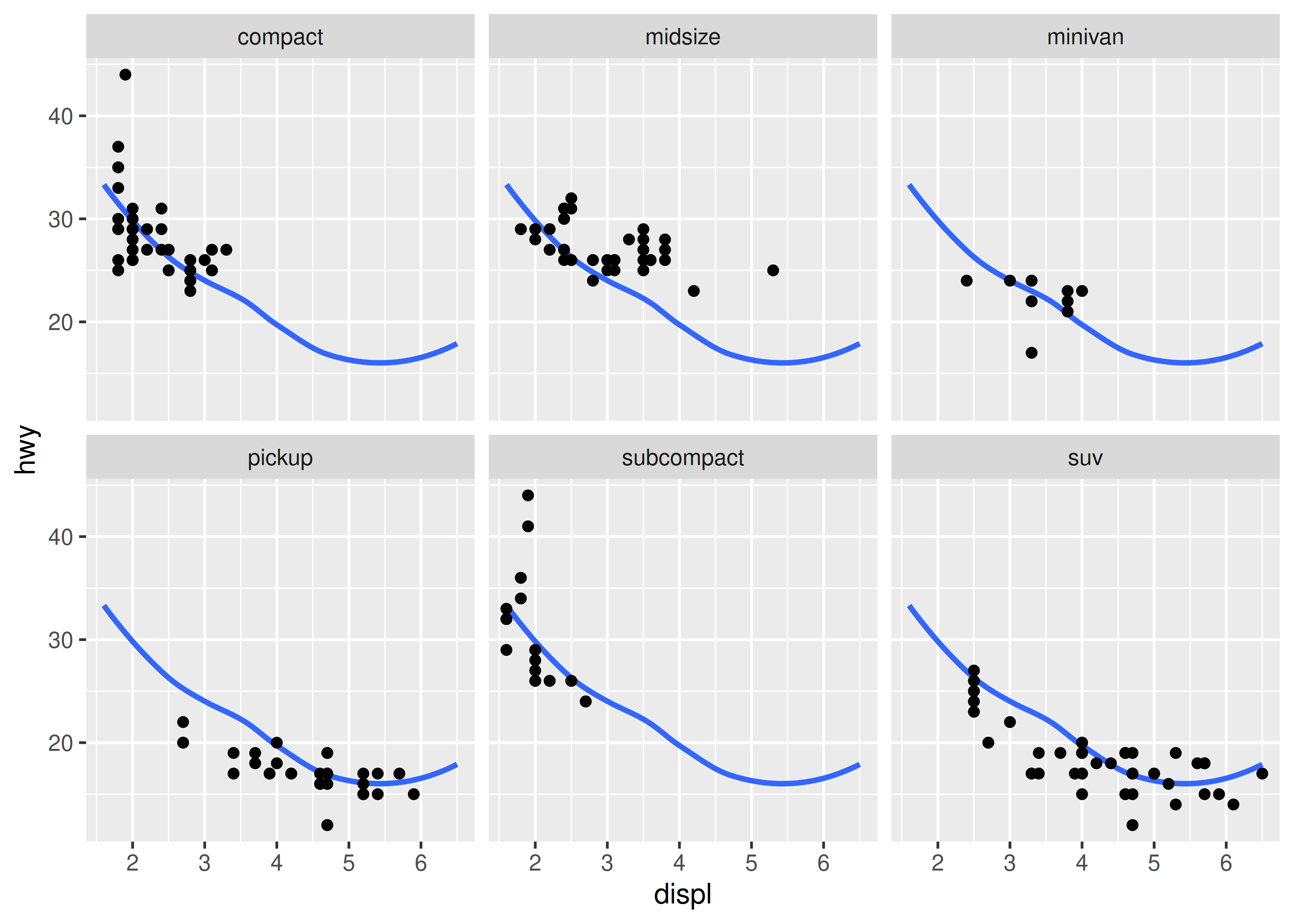
Recrea la siguiente gráfica. Faceta mpg2 por clase, superponiendo una curva suave que se ajusta al conjunto de datos completo.
#> `geom_smooth()` using method = 'loess' and formula = 'y ~ x'